(1.国家食品安全风险评估中心 卫生部食品安全风险评估重点实验室,北京100021; 2.北京农学院食品科学与工程学院,北京102206; 3.中国疾病预防控制中心 传染病预防控制所,北京102206)
作者简介: 彭子欣女副研究员研究方向为食品微生物学E-mail:pengzixin@cfsa.net.cn
通信作者: 李凤琴女研究员研究方向为食品微生物学E-mail:lifengqin@cfsa.net.cn
收稿日期: 2017-03-13
基金项目: 国家自然科学基金(31601574);中国博士后基金(2016M590072);北京青年拔尖人才(2014000021223ZK46);北京市科技新星交叉学科合作项目(Z161100004916029)
interaction relationships with host bacterium
(1.Key Laboratory of Food Safety Risk Assessment of Ministry of Health,China National Center for Food Safety Risk Assessment,Beijing 100021,China; 2.Food Science and Engineering College, Beijing University of Agriculture,Beijing 102206,China; 3.National Institute for Communicable Disease Control and Prevention,Chinese Center for Disease and Prevention,Beijing 102206,China)
肠球菌广泛存在于人体、动物、植物、土壤、水和食品中,目前细菌分类学上已将肠球菌划分为近50个种[3]。作为乳酸菌,肠球菌常被用于食品发酵,改善食品风味和延长货架期。而且,肠球菌还是一种益生菌,可用于改善人畜免疫系统,维持肠道菌群平衡[4]。然而,近几十年来发现,肠球菌也是社区和医院获得性感染的重要病原菌,常具有多重耐药性,可引起心内膜炎、菌血症、盆腔炎、新生儿感染、尿道炎等多种疾病[5]。有报道[1-2]显示,医院内高致病风险的粪肠球菌克隆群CC2携带有较多量的前噬菌体,其在促进菌株医院内适应性进化方面发挥了重要作用。属于CC2克隆群的粪肠球菌致病株V583基因组中的多个前噬菌体对宿主菌的致病性和耐药性有重要贡献,并且导致了人类心内膜炎感染。
希拉肠球菌是一种可引起人畜共患病的肠球菌。近年来,临床上由希拉肠球菌引起的急重症感染多有报道[6-8],但是对于希拉肠球菌的基因组信息还了解甚少[9-11],尤其是希拉肠球菌基因组中前噬菌体结构特征鲜有报道。前期工作中,已经从北京市市售生鲜猪肉中分离出1株达托霉素耐药希拉肠球菌R17,并对这株菌的全基因组开展了测序研究[12-14]。本研究将分析希拉肠球菌R17基因组中前噬菌体的分布及其结构特征,探讨前噬菌体与其宿主菌的毒力基因和耐药基因之间的进化关系,以期对前噬菌体与其食源性宿主菌的相互影响有进一步认识。
希拉肠球菌R17基因组上4个前噬菌体的起始位点和终止位点信息如表1所示。在4个噬菌体中,Prophage-3最长,达到35.7 kb,染色体上的3个噬菌体总长为66.0 kb,占染色体全长(2 886.5 kb)的2.29%。质粒上的Prophage-p长达35.2 kb,占到质粒全长(73.6 kb)的47.83%。Prophage-1的 GC含量(38.57%)高于染色体GC含量(36.96%),Prophage-2和Prophage-3的GC含量低于染色体的GC含量,其中Prophage-2的GC含量最低,为33.68%,Prophage-p的GC含量(36.24%)高于质粒的GC含量(35.57%)。
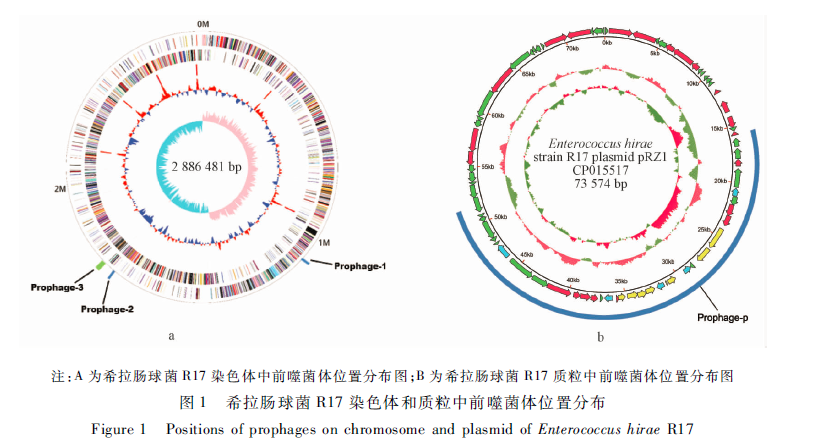
|
注:A为希拉肠球菌R17染色体中前噬菌体位置分布图;B为希拉肠球菌R17质粒中前噬菌体位置分布图 图1希拉肠球菌R17染色体和质粒中前噬菌体位置分布 Figure 1Positions of prophages on chromosome and plasmid of Enterococcus hirae R17 |
|
|
表1希拉肠球菌R17基因组中前噬菌体的分布及特征 Table 1Distribution and characteristics of prophages in the genome of Enterococcus hirae R17 注:括号中的数字表示噬菌体完整性评分;GC含量指鸟嘌呤和胞嘧啶所占比例 |
在CARD抗生素抗性基因数据库、VFDB毒力基因数据库和BacMet环境杀菌剂和重金属抗性基因数据库中预测发现,定位于染色体上的Prophage-1、Prophage-2和Prophage-3中不含有抗生素抗性基因、毒力基因、环境杀菌剂抗性基因和重金属抗性基因。
Prophage-p中含有13个编码序列,5个噬菌体附着att序列(见图2和表5)。13个编码序列中编码前噬菌体基因8个,其中3个编码转座酶蛋白。编码细菌基因的CDS有5个,分别编码红霉素抗性基因、杆菌肽抗性基因和噬菌体流产感染蛋白。

|
图2希拉肠球菌基因组中前噬菌体编码基因示意图 Figure 2Coding genes of prophages in the genome of Enterococcus hirae R17 |

|
表2希拉肠球菌R17染色体中前噬菌体Prophage-1基因特征 Table 2Gene characteristics of Prophage-1 in the Enterococcus hirae R17 chromosome 注:—表示假设蛋白;NA表示功能未知 |
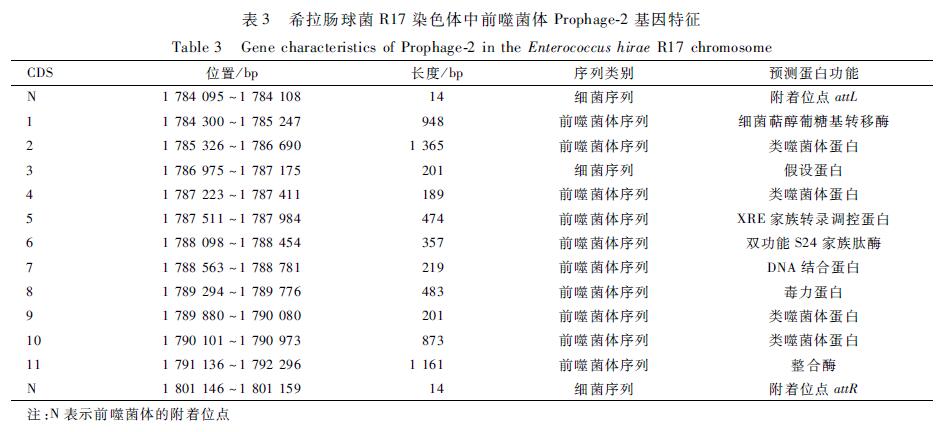
|
表3希拉肠球菌R17染色体中前噬菌体Prophage-2基因特征 Table 3Gene characteristics of Prophage-2 in the Enterococcus hirae R17 chromosome 注:N表示前噬菌体的附着位点 |
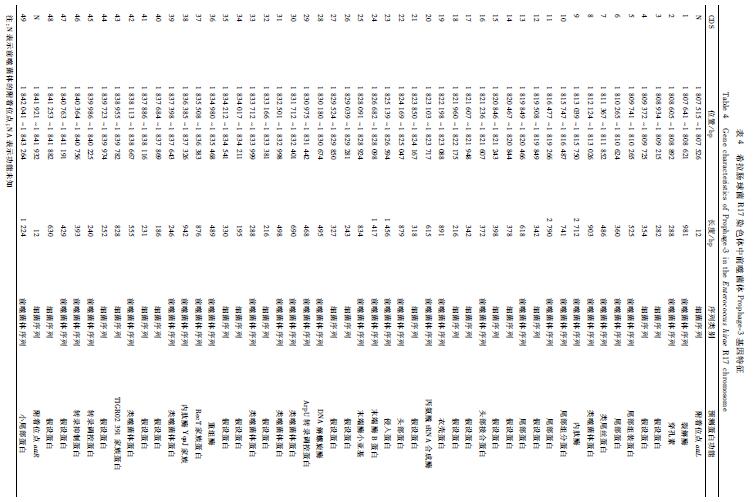
|
表4希拉肠球菌R17染色体中前噬菌体Prophage-3基因特征 Table 4Gene characteristics of Prophage-3 in the Enterococcus hirae R17 chromosome 注:N表示前噬菌体的附着位点;NA表示功能未知 |
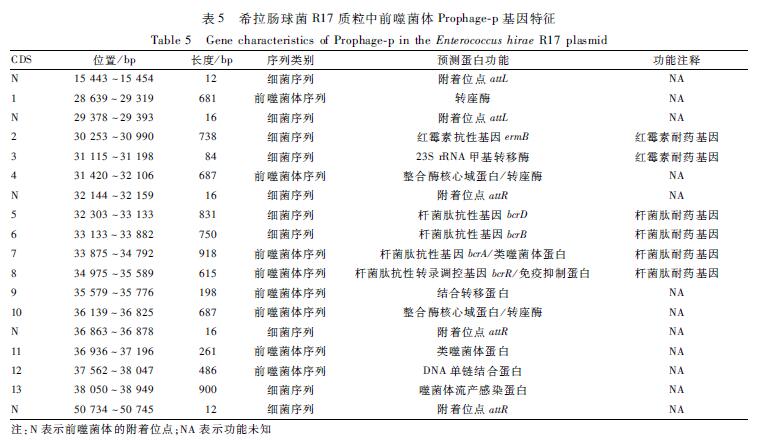
|
表5希拉肠球菌R17质粒中前噬菌体Prophage-p基因特征 Table 5Gene characteristics of Prophage-p in the Enterococcus hirae R17 plasmid 注:N表示前噬菌体的附着位点;NA表示功能未知 |
对多株细菌的基因组开展测序分析发现,前噬菌体是细菌短期内表现出种内或种间生物多样性的主要原因[20]。临床分离致病株粪肠球菌V583基因组中含有7个完整的前噬菌体,序列总长超过染色体大小的25%[1]。尽管Prophage-1、Prophage-2、Prophage-3仅占希拉肠球菌R17染色体总长的2.29%,Prophage-p仅占质粒全长的47.83%,但Prophage-2、Prophage-3和Prophage-p序列与肠球菌属种内和种间序列的覆盖度都较低,最低为39%。这不仅说明前噬菌体是造成肠球菌种内和种间基因分化的主要动力,也说明了希拉肠球菌R17前噬菌体中曾普遍发生过基因删除和重组事件。
前噬菌体与细菌的致病力、耐药性以及环境抗性也有重要关系[20]。细菌基因组中携带的前噬菌体往往是造成菌株之间致病性差异的主要原因。肠球菌是人体肠道常驻菌群的重要组成部分,但是致病株粪肠球菌V583基因组中就携带了前噬菌体编码的类血小板结合蛋白(platelet-binding-like protein),这种蛋白可黏附人类血小板,造成人类菌血症感染[2]。近年来有研究[2]发现,在很多粪肠球菌的临床分离株的基因组中都发现了多聚前噬菌体现象(polylysogeny),这说明前噬菌体在增加致病株的医院环境适应性方面发挥着重要作用。尽管在希拉肠球菌R17的前噬菌体中没有发现毒力基因,但应注意前噬菌体导致的菌株致病性变化。
噬菌体是造成抗生素耐药性快速扩散的主要原因之一。近年来研究[2]发现,氟喹诺酮类抗生素可以增强噬菌体的活性,导致细菌耐药性的加速传播。对奥地利市售50份鸡肉样品进行分析,发现有近一半样品遭到了噬菌体污染,而这种噬菌体可以有效地将抗生素耐药基因在细菌之间进行转移[21]。本研究发现,市售生鲜猪肉中分离的希拉肠球菌R17的红霉素抗性、杆菌肽抗性都是由Prophage-p介导的,这说明前噬菌体在增加菌株多重耐药性方面发挥了关键作用。由于噬菌体对于食品工业常用消毒剂如酒精等,具有极强的耐受性,提示应密切关注噬菌体导致的耐药基因在食源性细菌中的扩散,合理采取消毒措施。
噬菌体作为应对抗生素抗性蔓延的重要武器,也引起了广泛关注。由于噬菌体抗细菌感染的高特异性以及对人体的低毒性,可以减少抗生素的使用,一些前苏联国家长期使用噬菌体疗法对抗细菌感染类疾病[22],因此,研究细菌和噬菌体的相互影响,不仅可以深入认识细菌尤其是致病菌的致病性、耐药性和环境适应性的分子机制,而且对于监控致病菌变异、开发新型无抗生素抗菌疗法具有重要意义。
[2]MATOS R C, LAPAQUE N, RIGOTTIER-GOIS L, et al. Enterococcus faecalis prophage dynamics and contributions to pathogenic traits[J]. PLoS Genet, 2013, 9(6):e1003539.
[3]ABRIOUEL H, OMAR N B, MOLINOS A C, et al. Comparative analysis of genetic diversity and incidence of virulence factors and antibiotic resistance among enterococcal populations from raw fruit and vegetable foods, water and soil, and clinical samples[J]. Int J Food Microbiol, 2008, 123(1/2):38-49.
[4]BONACINA J, SUAREZ N, HORMIGO R, et al. A genomic view of food-related and probiotic Enterococcus strains[J]. DNA Res, 2016, 24(1):11-24.
[5]VAN S W, TOP J, RILEY D R, et al. Pyrosequencing-based comparative genome analysis of the nosocomial pathogen Enterococcus faecium and identification of a large transferable pathogenicity island[J]. BMC Genomics, 2010, 11(1):239-256.
[6]GHOSH A, BORST L, STAUFFER S H, et al. Mortality in kittens is associated with a shift in ileum mucosa-associated enterococci from Enterococcus hirae to biofilm-forming Enterococcus faecalis and adherent Escherichia coli[J]. J Clin Microbiol, 2013, 51(11):3567-3578.
[7]ANGHINAH R, WATANABE R G, SIMABUKURO M M, et al. Native valve endocarditis due to Enterococcus hirae presenting as a neurological deficit[J]. Case Rep Neurol Med, 2013:636070.
[8]BOURAFA N, LOUCIF L, BOUTEFNOUCHET N, et al. Enterococcus hirae, an unusual pathogen in humans causing urinary tract infection in a patient with benign prostatic hyperplasia: first case report in Algeria[J]. New Microbes New Infect, 2015, 8: 7-9.
[9]GAECHTER T, WUNDERLIN C, SCHMIDHEINI T, et al. Genome sequence of Enterococcus hirae (Streptococcus faecalis) ATCC 9790, a model organism for the study of ion transport, bioenergetics, and copper homeostasis[J]. J Bacteriol, 2012, 194(18): 5126-5127.
[10]PORCELLATO D, OSTLIE H M, SKEIE S B. Draft genome sequence of Enterococcus hirae strain INF E1 isolated from cultured milk[J]. Genome Announc, 2014, 2(4):e00948.
[11]KATYAL I, CHABAN B, HILL J E. Comparative genomics of cpn60-defined Enterococcus hirae ecotypes and relationship of gene content differences to competitive fitness[J]. Microb Ecol, 2016, 72(4): 917-930.
[12]彭子欣, 王伟, 胡豫杰, 等. 食品及环境样品中肠球菌快速检验方法的建立及优化[J]. 食品安全质量检测学报, 2016, 7(6): 2240-2246.
[13]彭子欣, 张爽, 王伟, 等. 北京市集贸市场生鲜猪肉肠球菌的耐药特征分析[J]. 中国食品卫生杂志, 2016, 28(6):736-743.
[14]PENG Z X, WANG W, HU Y J, et al. Complete genome sequence of Enterococcus hirae R17, a daptomycin-resistant bacterium isolated from retail pork in China[J]. Genome Announc, 2016, 4(3):606-616.
[15]ZHOU Y, LIANG Y J, LYNCH K, et al. PHAST: a fast phage search tool[J]. Nucl Acids Res, 2011, 39(suppl 2): 347-352.
[16]MCARTHUR A G, WAGLECHNER N, NIZAM F, et al. The comprehensive antibiotic resistance database[J]. Antimicrob Agents Ch, 2013, 57(7): 3348-3357.
[17]CHEN L H, ZHENG D D, LIU B, et al. VFDB 2016: hierarchical and refined dataset for big data analysis—10 years on[J]. Nucleic Acids Res, 2016, 44: 694-697.
[18]PAL C D, BENGTSSON-PALME J, RENSING C, et al. BacMet: antibacterial biocide and metal resistance genes database[J]. Nucleic Acids Res, 2014, 42: 737-743.
[19]蔡刘体, 刘艳霞, 孟琳, 等. 烟草青枯菌FQY_4基因组中原噬菌体生物信息学分析[J]. 中国烟草学报, 2015, 21(1):82-88.
[20]黎庶, 胡福泉. 前噬菌体[J]. 微生物通报, 2009, 36(3): 432-438.
[21]SHOUSHA A, AWAIWANONT N, SOFKA D, et al. Bacteriophages isolated from chicken meat and the horizontal transfer of antimicrobial resistance genes[J]. Appl Environ Microbiol, 2015, 81(14):4600-4606.
[22]REARDON S. Phage therapy gets revitalized[J]. Nature, 2014, 510(7503):15-16.
 彭子欣,张思雨,闫韶飞,王伟,王帅,甘辛,张建中,李凤琴.食源性希拉肠球菌R17基因组中前噬菌体的结构特征及其与宿主菌的相互影响[J].中国食品卫生杂志,2017,29(4):393-399.
彭子欣,张思雨,闫韶飞,王伟,王帅,甘辛,张建中,李凤琴.食源性希拉肠球菌R17基因组中前噬菌体的结构特征及其与宿主菌的相互影响[J].中国食品卫生杂志,2017,29(4):393-399.