DOi:10.13590/j.cjfh.2017.05.002
利用recA基因序列分析鉴定云南酵米面中伯克霍尔德菌属分离株
(1.武汉轻工大学生物与制药工程学院,湖北 武汉 430023;2.云南省疾病预防控制中心 食品安全与营养研究中心,云南 昆明 650022)
黄庭轩 男 本科生 研究方向为微生物 E-mail:htx0625@163.com
许燕 女 副主任技师 研究方向为卫生检验与食品安全 E-mail:286392468@qq.com
收稿日期:2017-06-07
云南省卫生科技计划项目(2014NS351);武汉轻工大学大学生科研项目(xsky2017008)
摘要: 目的 利用recA基因序列分析对云南省两起酵米面食物中毒案例中伯克霍尔德菌属病原菌进行分类鉴定。方法 对基于16S rRNA序列分析鉴定为伯克霍尔德菌属的4株分离菌株和1株唐菖蒲伯克霍尔德菌标准株(CICC 10574),通过聚合酶链式反应(PCR)扩增recA基因片段、测序,将测序结果与GenBank中伯克霍尔德菌属中73个种的对应片段序列比对,并构建系统发育树。结果 比较分析伯克霍尔德菌属中73个种的recA基因的亲缘关系,recA基因可以准确地区分鉴定伯克霍尔德菌属中包括食物中毒案例中4个分离株的不同的种,特别是在区分唐菖蒲伯克霍尔德种内不同致病亚种时有参考价值。结论 recA基因序列分析将酵米面食物中毒分离株鉴定为唐菖蒲伯克霍尔德菌,而且食物中毒案例中的4个分离株和植物病原菌及环境分离株的唐菖蒲伯克霍尔德菌的基因序列不同,具有独特的单核苷酸多态性(SNP),建议鉴定结果采用唐菖蒲伯克霍尔德菌椰毒致病变种(Burkholderia gladioli pv. cocovenenans)。
关键词:
酵米面; recA序列分析; 食物中毒; 唐菖蒲伯克霍尔德菌; 唐菖蒲伯克霍尔德菌椰毒致病变种; 食源性致病菌
中图分类号: R155 文献标识码:A 文章编号:1004-8456(2017)05-0526-08
Identification of isolates in fermented corn flour food poisoning case in China
by recA sequence analysis
by recA sequence analysis
(1.School of Biological and Pharmaceutical Engineering,Wuhan Polytechnic University, Hubei Wuhan 430023,China; 2.Yunnan Center of Disease Control and Prevention,Yunnan Kunming 650022,China)
Abstract:Objective The four pathogens isolated from two outbreaks caused by fermented corn flour products in Yunnan Province were identified by recA partial sequence analysis. Methods The four isolates were already identified as Burkholderia spp. on the basis of 16S rRNA sequence analysis. They were amplified by recA specific primers in polymerase chain reaction together with Burkholderia gladioli (CICC 10574). The products were sequenced. The sequences were blasted with 73 species in Burkholderia genus and a phylogenetic tree was constructed. Results Comparative analysis of genetic relationship of recA gene could identify and classify the 73 species of Burkholderia, even subspecies of B.gladioli. Conclusion The four isolates from food poisoning cases could be identified as B.gladioli by recA sequence analysis. Their sequences were different from those of plant pathogens of B.gladioli with specific single nucleotide polymorphism(SNP), and was identified as Burkholderia gladioli pv.cocovenenans.
Key words:
Fermented corn flour; recA sequence analysis; food poisoning; Burkholderia gladioli; Burkholderia gladioli pv.cocovenenans; foodborne pathogenic bacteria
变质银耳及自制淀粉类发酵食品引起的食物中毒曾是我国病死率最高的细菌性食物中毒之一,国标称其病原菌为椰毒假单胞菌(Pseudomonas cocovenenans)或椰毒假单胞菌酵米面亚种(Pseudomonas cocovenenans subsp. farinofermen-tans)[1-2]。2012和2014年在云南省红河州和文山州发生的两起酵米面(吊浆粑)食物中毒死亡案例的流行病学特征和毒素分析均和椰毒假单胞菌食物中毒一致,但VITEK全自动微生物分析系统鉴定其分离株为唐菖蒲伯克霍尔德菌(Burkholderia gladioli),并有5个生理生化指标与GB/T 4789.29—2003《食品卫生微生物学检验 椰毒假单胞菌酵米面亚种检验》不同[3-4]。经典生理生化鉴定法有其局限性,故采用分子生物学方法对分离株进行16S rRNA序列分析,将其鉴定到伯克霍尔德菌属(Burkholderia spp.)并与唐菖蒲伯克霍尔德菌最近源,但因该序列保守性鉴定到种存在争议[3-5]。 目前伯克霍尔德菌属有90个种,具有复杂多样性,既有人畜共患病原菌,也有植物病原菌及环境分离株。该属绝大多数种无致病力,其生态位与植物密切相关,属于根圈细菌或植物内生菌,甚至能促进植物生长或抗逆性。某些种能降解原油、除草剂和合成污染物等难降解化合物,但也包括部分重要植物致病菌,能感染多种植物宿主,造成重要农作物大幅减产,如水稻细菌性谷枯病菌(Burkholderia glumae)能感染水稻、番茄、芝麻、辣椒、茄子、紫苏等20多种植物导致枯萎病。该属中少数种是著名的人畜病原菌,如在澳大利亚和东南亚部分地区类鼻疽伯克霍尔德菌(Burkholderia pseudomallei)通过土壤或水接触伤口、消化系统或呼吸系统引发人畜的类鼻疽病急性或慢性感染。而与其近源的鼻疽伯克霍尔德菌(Burkholderia mallei)则可以导致马、驴或骡子的鼻疽病,偶尔也能通过这些动物传染给人类。两者都曾在战争中用做生物武器。而洋葱伯克霍尔德菌群(Burkholderia cepacia complex)对囊肿性纤维化(cystic fibrosis,CF)患者可引起严重危及生命的肺部感染[6]。 唐菖蒲伯克霍尔德菌种内多样性也非常复杂,国外研究[6]显示其在自然界广泛分布于土壤、水体、根圈和多种动物等生态位,常常与植物和真菌共生。该种也是著名的植物病原菌,根据侵染宿主被分为3个病原变种/型(pathovars):唐菖蒲伯克霍尔德菌唐菖蒲致病变种(Burkholderia gladioli pv. gladioli)、唐菖蒲伯克霍尔德菌洋葱致病变种(Burkholderia gladioli pv.alliicola)和唐菖蒲伯克霍尔德菌蘑菇致病变种(Burkholderia gladioli pv. agaricicola)分别导致唐菖蒲、洋葱和蘑菇的腐烂病。近年来,有研究[7]发现其与医院感染、囊性纤维化、角膜炎、骨髓炎肺部感染有关,尤其是在免疫力缺陷/受抑制人群中;2012年又有研究[8]发现它与新生儿的败血症和幼儿在医院感染的败血症有关;同时原椰毒假单胞菌也已并入唐菖蒲伯克霍尔德菌,有建议命名为唐菖蒲伯克霍尔德菌椰毒致病变种(Burkholderia gladioli pv.cocovenenans)[9],所以该种内也包括人类食源性致病菌。 随着分子生物学鉴定手段的普及,研究者也曾试图用分子生物学方式鉴定、区分该食源性致病菌,但是唐菖蒲伯克霍尔德菌椰毒致病变种不仅和唐菖蒲伯克霍尔德菌的3个植物致病变种在16S rRNA全长序列上差异很小,就连临近的植物伯克霍尔德菌和水稻细菌性谷枯病菌也很难区分[10]。所以本研究采用另一个管家基因recA序列分析对云南省酵米面食物中毒致死案例中4株分离株在伯克霍尔德菌属中的分类鉴定进行研究。recA是原核生物同源重组的中心分子,在DNA重组和DNA损伤应急修复(SOS)过程中是一个关键的基因,常用于做细菌分型,经研究[11-12]表明,recA基因可用于包括根癌农杆菌属、紫色色杆菌属等多种细菌的分型,已经有两项研究[13-14]证实其在整个伯克霍尔德属内以及在洋葱伯克霍尔德菌群的分型鉴定中有效。本研究试图采用recA序列分析来明确我国云南省近五年内两起酵米面食物中毒案例中分离株在目前微生物分类系统中的地位并探索该类食源性致病菌的新鉴定方法。
注:#表示该菌株为典型株(type strain);&表示全基因组测序完整图/草图;表中B.为Burkholderia属名缩写,P.为Paraburkholderia属名缩写
2×Taq PCR Master Mix 25 μl,正反引物各2 μl,DNA模板5 μl,加水至50 μl。利用Primer 3 Plus根据美国国家生物技术信息中心(NCBI)基因数据库recA序列(DQ989504.1~DQ989518.1)自行设计recA引物,recA-F:GATAGCAAGAAGGGCTCC,recA-R:CTCTTCTTCGTCCATCGCCTC。扩增条件为:94 ℃预变性5 min;94 ℃变性30 s,59 ℃退火30 s,72 ℃延伸45 s,循环30次;72 ℃延伸5 min。PCR产物长度为512 bp,用1%的琼脂糖凝胶电泳分离,Goldview染色,凝胶成像分析仪观察PCR扩增结果,并进行双向测序。
2014年加拿大学者[15]通过表型和比较基因组分析,将原伯克霍尔德菌属分为包含各种动物、人类、植物致病菌的伯克霍尔德菌属和一个含多种环境细菌的副伯克霍尔德菌新属,该结论已被一些机构所接受,所以表1中所选用的伯克霍尔德菌菌株中已有一些细菌属名改为副伯克霍尔德菌。该文献中采用21个保守蛋白质截短序列分析做出的进化树将伯克霍尔德菌属分为分支I和分支II两大类,而本研究采用的recA构建的进化树与其组成基本一致,说明recA序列分析对该属内各种鉴定非常有效,接近全基因组分析的结果。 在采用16S rRNA序列分析将两个案例中的4株分离株鉴定到伯克霍尔德菌属之后,本研究采用recA序列分析比较了伯克霍尔德菌属中72种不同种标准株的序列,以及唐菖蒲伯克霍尔德菌种内4个致病变种及环境分离株的recA序列分析,发现recA序列分析不仅能很好鉴别伯克霍尔德菌属内不同的种,对唐菖蒲伯克霍尔德菌种内变种的区分也有参考价值。
1 材料与方法
1.1 材料
1.1.1 菌株和序列来源
4株酵米面食物中毒事件分离株由云南省疾病预防控制中心保存[3-4],唐菖蒲伯克霍尔德菌标准株(CICC 10574)购自中国工业微生物菌种保藏管理中心,其他参比序列下载于GenBank数据库,详见表1。其中4个分离株经16S rRNA序列分析鉴定为伯克霍尔德菌属[3]。
1.1.2 主要仪器与试剂
Cycler PCR仪(美国Bio-Rad)、凝胶图像分析系统(英国SYNGENE)、DYY8B型稳压电泳仪、离心机、Nanopure纯水机。 马铃薯葡萄糖琼脂(PDA)培养基(英国OXOID),Premix Taq、DL2000 DNA Marker和核酸染料Goldview均购自日本TaKaRa,琼脂糖(西班牙Biowest),聚合酶链式反应(PCR)引物合成以及产物测序由金唯智科技有限公司完成。
1.2 方法
1.2.1 分离株的recA基因PCR扩增及测序
用PDA平板划线分离菌株,于30 ℃培养24 h,选取单菌落至装有50 μl无菌MiliQ水的PCR管中,轻振管壁使菌体在水中混匀,于98 ℃下高温裂解10 min,12 000 r/min离心5 min,所得上清液即为细菌DNA,转移至无菌EP管中,4 ℃保存备用。以recA为引物进行PCR扩增,PCR扩增体系: 注:#表示该菌株为典型株(type strain);&表示全基因组测序完整图/草图;表中B.为Burkholderia属名缩写,P.为Paraburkholderia属名缩写
2×Taq PCR Master Mix 25 μl,正反引物各2 μl,DNA模板5 μl,加水至50 μl。利用Primer 3 Plus根据美国国家生物技术信息中心(NCBI)基因数据库recA序列(DQ989504.1~DQ989518.1)自行设计recA引物,recA-F:GATAGCAAGAAGGGCTCC,recA-R:CTCTTCTTCGTCCATCGCCTC。扩增条件为:94 ℃预变性5 min;94 ℃变性30 s,59 ℃退火30 s,72 ℃延伸45 s,循环30次;72 ℃延伸5 min。PCR产物长度为512 bp,用1%的琼脂糖凝胶电泳分离,Goldview染色,凝胶成像分析仪观察PCR扩增结果,并进行双向测序。
1.2.2 recA序列分析
所得序列提交至GenBank数据库获得序列编号,并从数据库中提取伯克霍尔德菌属(包括伯克霍尔德菌属和副伯克霍尔德属Paraburkholderia)72个种的代表菌株recA序列和21个唐菖蒲伯克霍尔德菌种内菌株recA序列,用MEGA6.0软件对recA基因进行分析。
1.2.3 单核苷酸的多态性(SNP)分析
recA序列:以B.gladioli pv.cocovenenans LMG11626为参比序列,利用Clustalw软件对21个唐菖蒲伯克霍尔德菌种内菌株和最近源2个种的菌株进行recA序列比对,完成SNP分析。 16S rRNA序列:从GenBank网站下载唐菖蒲伯克霍尔德菌以及邻近菌株的16S rRNA序列,并利用DNAstar组件Megalign计算序列间的差异性。
2 结果
2.1 PCR扩增结果
以自行设计的引物recA-F和recA-R对4株食物中毒分离株和标准菌株(CICC 10574)同时进行PCR扩增,扩增效果良好,阴性对照未出现条带,5株菌株都有单一清晰条带,产物大小符合预期512 bp,见图1。PCR产物进行双向测序,测序结果提交GenBank获得编号(KY312856~KY312860)。sp;

|
注:1为Marker DL2000;2为空白对照;3为食物中毒分离株YD-1; 4为食物中毒分离株YD-2;5为食物中毒分离株YD-4;6为食物中毒分离株YD-HH;7为标准菌株(CICC 10574) 图1 分离株的recA基因扩增产物电泳图 Figure 1 Electropherogram of PCR amplification products of the recA gene |
2.2 recA序列分析构建进化树
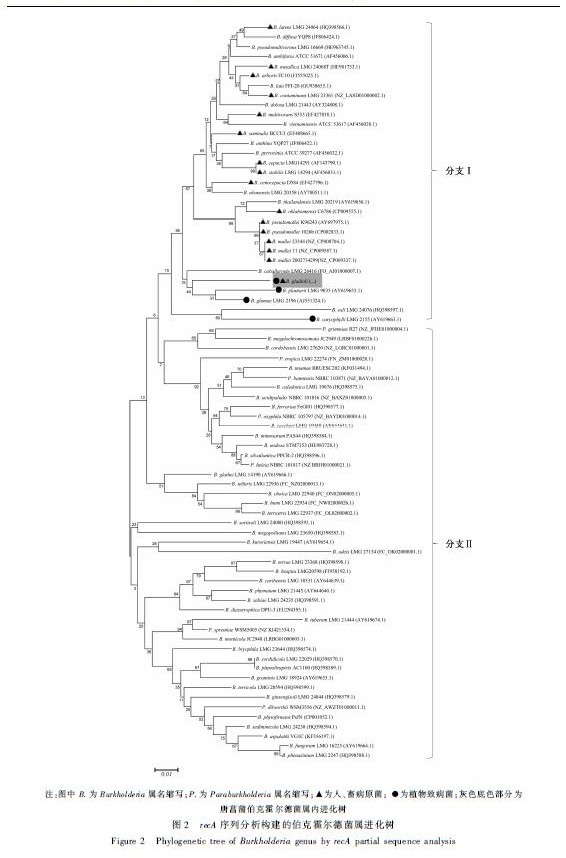
在GenBank数据库中选取唐菖蒲伯克霍尔德菌种内所有菌株及伯克霍尔德菌属其他72个种的菌株recA序列信息,详见表1,以这些recA序列比对构建的伯克霍尔德菌属进化树,见图2,其中唐菖蒲伯克霍尔德菌种内进化树见图3。 从表1可以看出GenBank数据库中伯克霍尔德菌属内菌株主要有环境分离株、植物致病菌和人/动物致病株,而单就唐菖蒲伯克霍尔德菌这一个种内就有环境分离株如CICC 10574和UAPS07070,植物病原菌唐菖蒲伯克霍尔德菌唐菖蒲致病变种、唐菖蒲伯克霍尔德菌洋葱致病变种、唐菖蒲伯克霍尔德菌蘑菇致病变种(数据库中目前没有该变种的recA序列)以及人类的食源性致病菌唐菖蒲伯克霍尔德菌椰毒致病变种。 由图2可见,目前GenBank数据库中所有唐菖蒲伯克霍尔德菌种菌株在recA序列分析构建的进化树上都位于同一个进化分支上,与该种最近源的都是植物病原菌,如伯克霍尔德菌属细菌中水稻细菌性谷枯病菌和水稻幼苗枯萎的病原菌植物伯克霍尔德菌。同时该种与伯克霍尔德菌属中人类条件致病菌洋葱伯克霍尔德菌群和人畜共患病原菌类鼻疽伯克霍尔德菌群(B.pseudomallei group)也近源。图2中所有命名为副伯克霍尔德属(Parabukholderia)的菌株和多数环境分离株都在分支II,而所有病原菌,不论是植物病原菌还是人类、动物致病株都集中于上部的分支I。 由图3可见,唐菖蒲伯克霍尔德菌种内以recA序列分析所构建的进化树中,来自云南不同地区的两起不同年份的食物中毒案例中的分离株YD-HH、YD-1、YD-2和YD-4在同一个进化分支上,它们与原椰毒假单胞菌典型株B.gladioli pv.cocovenenans LMG 11626近源;唐菖蒲伯克霍尔德菌种中另外2个植物病原菌变种唐菖蒲伯克霍尔德菌唐菖蒲致病变种、唐菖蒲伯克霍尔德菌洋葱致病变种都单独构成一个进化分支,分离自水果的两个环境分离株CICC 10574和UAPS07070在同一进化分支上,只有韩国分离的水稻病原菌B.gladioli BSR3较为独特单独成为一只分支。
2.3 唐菖蒲伯克霍尔德菌种内及植物伯克霍尔德菌recA序列的SNP比较
将唐菖蒲伯克霍尔德菌种内21株菌株的recA序列进行SNP比较,结果见图4。 对recA的416 bp长度序列的比较发现,来自云南不同地区的两起食物中毒案例中的分离株YD-HH、YD-1、YD-2和YD-4的序列完全一致(故图4中仅列出YD-HH),但是它们和分离自印尼爪哇由椰子发酵制备的天培而导致食物中毒案例的原椰毒假单胞菌的典型株B.gladioli pv.cocoven-enans LMG 11626之间有2 bp的差异。而GenBank数据库中所有的12个唐菖蒲伯克霍尔德菌洋葱致病变种菌株的序列也是完全一致的,所以仅以LMG 6979序列为代表。分离自植物的CICC 10574和UAPS07070序列一致,以CICC 10574为代表。唐菖蒲伯克霍尔德菌变种的典型株B.gladioli pv.gladioli LMG 2216和韩国分离的水稻病原菌B.gladioli BSR3序列独特则单独列出。图4可见,凡是唐菖蒲伯克霍尔德菌种内所有的菌株在recA基因416 bp的序列中仅有1~3 bp的微小差别,序列之间相似度高达99.28%~99.76%;但是它们和与其序列最相似的B.glumae LMG 2196之间差异达20 bp,相似度95.16%;与植物伯克霍尔德菌种标准株B.plantarii LMG 9035之间差异达28 bp,相似度93.27%。 同样比较这几个标准株1 124 bp的16S rRNA序列,唐菖蒲伯克霍尔德菌种内菌株相似度为99.4%~100.0%,而与B.glumae LMG 2196和B.plantarii LMG 9035相似度也高达98.8%和98.9%。可见recA序列比较能够准确充分地将唐菖蒲伯克霍尔德菌种和伯克霍尔德菌属的近源种区分开,甚至在唐菖蒲伯克霍尔德菌种内各变种区分上有参考价值。而16S rRNA序列则过于保守,很难将唐菖蒲伯克霍尔德菌种与近源的其他种明确地区分开。
|
注:图中B.为Burkholderia属名缩写;P.为Paraburkholderia属名缩写;▲为人、畜病原菌; ●为植物致病菌;灰色底色部分为 唐菖蒲伯克霍尔德菌属内进化树 注:图中B.为Burkholderia属名缩写;P.为Paraburkholderia属名缩写;●为植物致病菌 图2 recA序列分析构建的伯克霍尔德菌属进化树 Figure 2 Phylogenetic tree of Burkholderia genus by recA partial sequence analysis |

|
图3 recA序列分析构建的唐菖蒲伯克霍尔德菌种 内进化树 Figure 3 Phylogenetic tree of Burkholderia gladioli species by recA partial sequence analysis |
3 讨论
椰毒假单胞菌食物中毒曾是我国死亡率最高的细菌性食物中毒之一,遍及我国20多个省份[1],后经过卫生部门的大力宣传和消费者生活水平的提高,食用酵米面情况减少,这类食物中毒事件目前已经较为罕见,相关研究日见稀少。但2012—2014年云南省红河州和文山州2起唐菖蒲伯克霍尔德菌食物中毒事件病死率高达30.0%和55.6%,在社会上造成恶劣影响,经济损失巨大[3-4],所以食品安全监管部门不应放松对该类食源性致病菌的监控和研究。椰毒假单胞菌在国际分类系统中已经并入唐菖蒲伯克霍尔德菌,我国目前尚未修订该项食品安全国家标准,细菌名称与国际不接轨,导致很多困扰。如云南省这两起食物中毒事件从流行病调查特征、毒素检测结果与GB/T 4789.29—2003椰毒假单胞菌食物中毒一致,生理生化鉴定和分子生物学鉴定都与唐菖蒲伯克霍尔德菌一致,建议在标准修订时对其名称进行更新。

|
注:黑底色为与标准菌株LMG11626序列差异位点 图4 唐菖蒲伯克霍尔德菌种内不同菌株之间recA序列的SNP比较 Figure 4 SNP analysis of recA sequences in Burkholderia gladioli species |
参考文献
[1] 刘秀梅. 我国椰毒假单胞菌酵米面亚种食物中毒流行趋势浅析[J]. 中华预防医学杂志,1996,30(6):372-374.
[2] 中华人民共和国卫生部,中国国家标准化管理委员会.食品卫生微生物学检验 椰毒假单胞菌酵米面亚种检验:GB/T 4789.29—2003[S]. 北京:中国标准出版社,2003.
[3] 周帼萍, 梁泉, 黄庭轩,等. 云南省文山州广南县吊浆粑食物中毒事件的病原学分析[J]. 中国食品卫生杂志, 2017,29(1):71-75.
[4] 杨庆文, 国译丹, 周惠新,等. 云南省首起唐菖蒲伯克霍尔德菌食物中毒的鉴定与分析[J]. 中国卫生检验杂志, 2013,23(17): 3363-3364,3367.
[5] 焦振泉, 刘秀梅, 杨瑞馥,等. 椰毒假单胞菌酵米面亚种16S rDNA序列测定与分析[J]. 卫生研究, 1999, 28(4):232-235.
[6] CHAIN P S G. Genomic versatility in the Burkholderia genus: from strains to species[D]. East Lansing: Doctoral Dissertation of Michigan State University, 2011.
[7] JONES A M, STANBRIDGE T N, ISALSKA B J, et al. Burkholderia gladioli: recurrent abscesses in a patient with cystic fibrosis[J]. Journal of Infection, 2001, 42(1):69-71.
[8] DURSUN A, ZENCIROGLU A, KARAGOL B S, et al. Burkholderia gladioli sepsis in newborns[J]. European Journal of Pediatrics, 2012, 171(10):1503-1509.
[9] JIAO Z Q,YOSHIAKI K,NORIKO M,et al. Need to differentiate lethal toxin-producing strains of Burkholderia gladioli, which cause severe food poisoning: description of B.gladioli pathovar cocovenenans and an emended description of B.gladioli[J]. Microbiology and Immunology, 2003, 47(12):915-925.
[10] LIU D Y. Molecular detection of foodborne pathogens[M]. USA:Taylor and Francis CRC Press, 2009:338-343.
[11] COSTECHAREYRE D, RHOUMA A, LAVIRE C, et al. Rapid and efficient identification of Agrobacterium species by recA allele analysis: Agrobacterium recA diversity.[J]. Microbial Ecology, 2010, 60(4):862-872. [12] SCHOLZ H C, WITTE A, TOMASO H, et al. Genotyping of Chromobacterium violaceum isolates by recA PCR-RFLP analysis[J]. Fems Microbiology Letters, 2005, 244(2):347-352.
[13] PAYNE G W, VANDAMME P, MORGAN S H, et al. Development of a recA gene-based identification approach for the entire Burkholderia genus[J]. Applied and Environmental Microbiology, 2005, 71(7):3917-3927. [14] CESARINI S, BEVIVINO A, TABACCHIONI S, et al. RecA gene sequence and multilocus sequence typing for species-level resolution of Burkholderia cepacia complex isolates[J]. Letters in Applied Microbiology, 2009, 49(5):580-588.
[15] SAWANA A, ADEOLU M, GUPTA R S. Molecular signatures and phylogenomic analysis of the genus Burkholderia: proposal for division of this genus into the emended genus Burkholderia containing pathogenic organisms and a new genus Paraburk-holderia gen. nov. harboring environmental species[J]. Front Genet,2014,5:429.
[2] 中华人民共和国卫生部,中国国家标准化管理委员会.食品卫生微生物学检验 椰毒假单胞菌酵米面亚种检验:GB/T 4789.29—2003[S]. 北京:中国标准出版社,2003.
[3] 周帼萍, 梁泉, 黄庭轩,等. 云南省文山州广南县吊浆粑食物中毒事件的病原学分析[J]. 中国食品卫生杂志, 2017,29(1):71-75.
[4] 杨庆文, 国译丹, 周惠新,等. 云南省首起唐菖蒲伯克霍尔德菌食物中毒的鉴定与分析[J]. 中国卫生检验杂志, 2013,23(17): 3363-3364,3367.
[5] 焦振泉, 刘秀梅, 杨瑞馥,等. 椰毒假单胞菌酵米面亚种16S rDNA序列测定与分析[J]. 卫生研究, 1999, 28(4):232-235.
[6] CHAIN P S G. Genomic versatility in the Burkholderia genus: from strains to species[D]. East Lansing: Doctoral Dissertation of Michigan State University, 2011.
[7] JONES A M, STANBRIDGE T N, ISALSKA B J, et al. Burkholderia gladioli: recurrent abscesses in a patient with cystic fibrosis[J]. Journal of Infection, 2001, 42(1):69-71.
[8] DURSUN A, ZENCIROGLU A, KARAGOL B S, et al. Burkholderia gladioli sepsis in newborns[J]. European Journal of Pediatrics, 2012, 171(10):1503-1509.
[9] JIAO Z Q,YOSHIAKI K,NORIKO M,et al. Need to differentiate lethal toxin-producing strains of Burkholderia gladioli, which cause severe food poisoning: description of B.gladioli pathovar cocovenenans and an emended description of B.gladioli[J]. Microbiology and Immunology, 2003, 47(12):915-925.
[10] LIU D Y. Molecular detection of foodborne pathogens[M]. USA:Taylor and Francis CRC Press, 2009:338-343.
[11] COSTECHAREYRE D, RHOUMA A, LAVIRE C, et al. Rapid and efficient identification of Agrobacterium species by recA allele analysis: Agrobacterium recA diversity.[J]. Microbial Ecology, 2010, 60(4):862-872. [12] SCHOLZ H C, WITTE A, TOMASO H, et al. Genotyping of Chromobacterium violaceum isolates by recA PCR-RFLP analysis[J]. Fems Microbiology Letters, 2005, 244(2):347-352.
[13] PAYNE G W, VANDAMME P, MORGAN S H, et al. Development of a recA gene-based identification approach for the entire Burkholderia genus[J]. Applied and Environmental Microbiology, 2005, 71(7):3917-3927. [14] CESARINI S, BEVIVINO A, TABACCHIONI S, et al. RecA gene sequence and multilocus sequence typing for species-level resolution of Burkholderia cepacia complex isolates[J]. Letters in Applied Microbiology, 2009, 49(5):580-588.
[15] SAWANA A, ADEOLU M, GUPTA R S. Molecular signatures and phylogenomic analysis of the genus Burkholderia: proposal for division of this genus into the emended genus Burkholderia containing pathogenic organisms and a new genus Paraburk-holderia gen. nov. harboring environmental species[J]. Front Genet,2014,5:429.
 黄庭轩,赵梦馨,周帼萍,许燕,杨祖顺.利用recA基因序列分析鉴定云南酵米面中伯克霍尔德菌属分离株[J].中国食品卫生杂志,2017,29(5):526-533.
黄庭轩,赵梦馨,周帼萍,许燕,杨祖顺.利用recA基因序列分析鉴定云南酵米面中伯克霍尔德菌属分离株[J].中国食品卫生杂志,2017,29(5):526-533.