DOi:10.13590/j.cjfh.2018.01.017
北京市市售牡蛎中诺如病毒污染对居民健康影响的初步定量风险评估
(国家食品安全风险评估中心 卫生部食品安全风险评估重点实验室,北京 100021)
收稿日期:2017-10-30
作者简介:宋筱瑜 女 副研究员 研究方向为食品微生物风险评估 E-mail:xiaoyu.song@cfsa.net.cn
通信作者:朱江辉 男 副研究员 研究方向为食品微生物风险评估 E-mail:zhujianghui@cfsa.net.cn
基金项目:北京市自然科学基金(5141002)
摘要:目的 通过建立针对北京市居民从零售到餐桌的牡蛎-诺如病毒暴露模型,对北京市居民通过市售牡蛎暴露诺如病毒的风险开展初步定量评估。方法 利用北京市市售牡蛎中诺如病毒的356条定量检测数据,模拟北京市居民牡蛎消费情景,建立暴露评估模型,结合文献发表的基于暴发数据推导的剂量-反应关系模型,对居民通过生食牡蛎后发生诺如病毒感染的健康风险及其影响因素进行评估。结果 GI型和GII型诺如病毒阳性样品的污染水平分别为2.62×104个病毒拷贝/g(95%CI:3.73×103~1.54×105)和5.02×104个病毒拷贝/g(95%CI:8.13×103~2.52×105)。对血清分泌受体阳性的人群,生食1个可能受GI型和GII型诺如病毒污染的牡蛎的发病风险分别为0.93(95%CI:0.73~0.98)和0.95(95%CI:0.80~0.99);对血清分泌受体阴性的人群,生食1个可能污染GI型和GII型诺如病毒牡蛎的发病风险分别为0.37(95%CI:0.04~0.64)和0.57(95%CI:0.07~0.99)。风险的估计值与诺如病毒阴性样品的赋值相关,相关系数为0.49。不确定性分析显示,现有检测方法的检出限高于半数感染及致病剂量,是评估结果不确定性的主要来源。结论 北京市居民通过生食牡蛎发生食源性诺如病毒感染的风险很高;提高病毒拷贝定量检测技术的灵敏度,降低检测方法的检出限水平是降低评估结果不确定性的主要途径。
关键词:
诺如病毒; 牡蛎; 健康风险; 风险评估; 食品安全
文章编号:1004-8456(2018)01-0079-05
中图分类号:R155
文献标志码:A
Preliminary quantitative risk assessment of Norovirus in oysters at retail for Beijing residents
(Key Laboratory for Food Safety Risk Assessment of Ministry of Health,China National Centerfor Food Safety Risk Assessment,Beijing 100021,China)
Abstract:Objective To conduct a quantitative risk assessment for Staphylococcus aureus in ready-to-eat salads, and seek effective preventive measures according to the risk assessment result.Methods Combining quantitative data of Staphylococcus aureus with residents’ consumption data of ready-to-eat salads, a quantitative risk assessment was performed according to the codex guidelines with four steps: hazard identification, hazard characterization, exposure assessment and risk characterization. A Monte Carlo simulation method was employed to analyze the health risk of Staphylococcus aureus by consuming ready-to-eat salads.Results The contamination rate of Staphylococcus aureus in ready-to-eat salads was 3.39% (21/620) and the average concentration was 229 CFU/g. The average intake was 56.27 times per year. The estimated probability of infectious illness caused by Staphylococcus aureus in ready-to-eat salads in Shanghai was 0.04%. That indicated the number of illness might reach 518.1 thousand cases every year.Conclusion There was certain risk of Staphylococcus aureus poisoning by consuming ready-to-eat salads for Shanghai residents. Decreasing initial contamination level and controlling the storage time and temperature were the critical approaches to reduce its risk.
Key words:
Vegetables; spatial statistics; pesticide residues; risk analysis
诺如病毒感染是人类胃肠炎的重要病因之一,全球每年因诺如病毒感染导致约250万人的伤残调整寿命年损失[1],美国诺如病毒感染导致了每年约550万例食源性病例的发生,占到已知病因的食源性疾病病例数的50%以上[2]。我国自1995年首次报道由诺如病毒引起的幼儿急性胃肠炎病例以来,全国各地相继报道诺如病毒引起的急性胃肠炎疫情[3]。流行病学研究[4-5]表明,诺如病毒胃肠炎暴发疫情,通常是从进食了受诺如病毒污染的食物或水开始,其中双壳贝类是诺如病毒食源性传播途径中的重要载体[6-7]。在国内外大多数由贝类引起的诺如病毒急性胃肠炎暴发中,最常见的贝类为牡蛎[8-9]。但是目前未见关于牡蛎中诺如病毒污染对居民健康影响的定量风险评估报告。
国家食品安全风险评估中心2014年对北京市部分地区零售环节牡蛎中诺如病毒污染水平开展了定量监测[10],基于此,本研究通过构建牡蛎-诺如病毒暴露评估模型,对不同人组织血清分泌受体的人群,通过生食可能被不同分型诺如病毒污染的牡蛎后产生的健康风险及其影响因素进行评估。

而对于再生食第2只牡蛎时,摄入的病毒可能量是第1只可能的剂量加上再随机选取第2只牡蛎时可能摄入的量。以此类推,若居民生食了20只牡蛎,摄入的病毒可能量是前19只牡蛎可能摄入的总剂量加上随机选取的第20只牡蛎中可能含有的病毒量。20只牡蛎是假设居民每餐可食入牡蛎的最大量,并且假设居民食入牡蛎均为整只食用,不考虑食用半只的情形。食用2~20只牡蛎可能摄入病毒的剂量,用公式(2)表示:
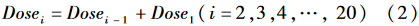
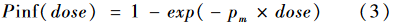 其中pm是贝塔分布输出结果,而dose是通过生食牡蛎摄入的诺如病毒粒子数的泊松分布结果,exp表示为指数函数。估计诺如病毒感染后的发病风险的公式如(4)所示:
其中pm是贝塔分布输出结果,而dose是通过生食牡蛎摄入的诺如病毒粒子数的泊松分布结果,exp表示为指数函数。估计诺如病毒感染后的发病风险的公式如(4)所示: 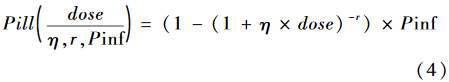 其中η和r是估计感染后发病概率公式的关键参数;四类人群的剂量-反应关系模型参数详见文献[12]。
其中η和r是估计感染后发病概率公式的关键参数;四类人群的剂量-反应关系模型参数详见文献[12]。
由于诺如病毒不能在体外进行培养,对于病毒在环境中沿食品链传递、转移等行为尚未有相关模型,特别是对于不同时间、温度等条件下烹调牡蛎可以导致的病毒的灭活模型尚未见相关报道,因此本评估中很难考虑经烹调后牡蛎中诺如病毒的存活量情况。我国居民牡蛎的消费普遍以白灼、炭烤等方式为主,但为了保留牡蛎的口感,这些烹调方式下中心温度可能只有60~70 ℃。该温度对牡蛎中病毒的灭活效力,尚无详实的研究报道,因此,在基于本次监测数据开展暴露评估时,模拟的是北京市居民生食牡蛎的情景下可能感染诺如病毒的风险。
根据THEBAULT等[12]的剂量-反应关系模型推算,Se+/GI和Se+/GII的半数感染剂量的中位数仅为7.1和1.6个病毒拷贝,而感染后致病的半数剂量的中位数仅为32和4.86个病毒拷贝。尽管本研究对牡蛎中诺如病毒的定量检测取得了较大的进展,但是检出限为每克消化腺100个基因拷贝,远远大于病毒的感染和致病剂量。由于目前对低于检出限的牡蛎中诺如病毒的污染密度未知,本次评估对阴性样品的取值用了均一分布,在0~100之间随机取值,其均值约在50个病毒拷贝,严重高估了人群的发病风险。根据敏感性分析结果可见,阴性样品的取值可极大影响最终发病风险的估计的准确性,这可能由于诺如病毒的感染和发病剂量极低,目前较高的病毒检出限难以定量估计最终的发病风险,因此迫切需要更为精确地病毒拷贝定量检测技术,获得低污染密度下的牡蛎污染情况,从而获得更为可靠的评估数据。研究结果提示,基于现有检测技术,生食诺如病毒检出阳性的牡蛎发生诺如病毒感染的风险非常高,建议居民尽可能避免生食牡蛎。收集诺如病毒随温度发生变化的相关数据,建立随牡蛎烹调温度变化而改变的诺如病毒动态模型,提高牡蛎中诺如病毒定量检测的敏感性,是进一步开展牡蛎中诺如病毒定量风险评估的重要数据支持和依据。
国家食品安全风险评估中心2014年对北京市部分地区零售环节牡蛎中诺如病毒污染水平开展了定量监测[10],基于此,本研究通过构建牡蛎-诺如病毒暴露评估模型,对不同人组织血清分泌受体的人群,通过生食可能被不同分型诺如病毒污染的牡蛎后产生的健康风险及其影响因素进行评估。
1材料与方法
1.1食品污染数据来源
2015年9月至2016年9月期间,江涛等[10]在北京市某海鲜批发市场采集新鲜牡蛎样品,分离消化腺,将2~5个牡蛎消化腺混合后为一份样品,共计356份样品。
1.2方法
1.2.1检测方法
用试剂盒提取病毒RNA,采用一步法实时荧光逆转录聚合酶链式反应(real time RT-PCR)检测样品中GI和GII基因组诺如病毒,并对阳性样品进行定量分析。
1.2.2暴露评估模型
本评估的暴露模型模拟的是北京市居民生食牡蛎的情景下感染诺如病毒的风险。通过生食一只牡蛎,可能摄入诺如病毒粒子的量可用公式表示为暴露量=牡蛎中诺如病毒浓度×牡蛎重量。由于牡蛎的检测用的是消化腺,获得的病毒浓度也是每克消化腺中病毒的含量。一般来说,每个牡蛎消化腺的重量约为1 g,则每克消化腺中病毒的含量就等于食用该牡蛎摄入的病毒数量。
1.2.3牡蛎中诺如病毒污染水平估计
假设以所有检测样品中诺如病毒污染的平均水平作为北京市居民消费牡蛎中诺如病毒的污染水平,为模拟不确定性,用贝塔分布描述牡蛎中诺如病毒的初始污染率(Pp),公式为Pp=Beta(s+1, n-s+1), 其中n表示检测的样品总数,s代表阳性样品数[11]。用累积分布描述阳性样品的污染水平(Cp),公式为Cp=Cumulative (min, max, {x1,x2,…, xn},{p1,p2,…, pn}),其中min和max分别为阳性样品检测值x1,x2,…, xn的最小值和最大值;p1,p2,…, pn为各检测值的累积概率。由于诺如病毒的检出限为100个基因拷贝,对于检测结果为阴性的样品,其污染水平用均一分布进行描述(Cn),公式为Cn=Uniform(min, max)[11],在此min认定为0,max认定为100。
1.2.4通过生食牡蛎暴露诺如病毒的剂量估计
对于居民每生食一只牡蛎是否会食用到被污染的牡蛎,该过程符合二项式分布,用Binomial(1,p)进行描述,其中p可以用牡蛎的阳性污染率Pp来表示。对于每生食一只牡蛎,摄入的诺如病毒可能剂量(Dose1)用公式(1)表示:
而对于再生食第2只牡蛎时,摄入的病毒可能量是第1只可能的剂量加上再随机选取第2只牡蛎时可能摄入的量。以此类推,若居民生食了20只牡蛎,摄入的病毒可能量是前19只牡蛎可能摄入的总剂量加上随机选取的第20只牡蛎中可能含有的病毒量。20只牡蛎是假设居民每餐可食入牡蛎的最大量,并且假设居民食入牡蛎均为整只食用,不考虑食用半只的情形。食用2~20只牡蛎可能摄入病毒的剂量,用公式(2)表示:
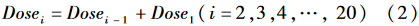
1.2.5评估方法
北京市居民通过双壳类水产品摄入诺如病毒的剂量估计采用Monte Carlo分析, 在@Risk软件6.0(Palisade Corporation)中迭代10 000次。抽样方法选择Latin Hypercube, 所生成的分布以平均值、中位数及第5和第95百分位数等方式表示。暴露评估的参数设置详见表1。|
|
表1牡蛎中诺如病毒评估模型的参数设置 Table 1Description of parameters in the exposure assessment model for the oysters-Norovirus combination |
1.2.6风险估计
根据THEBAULT等[12]通过暴发数据推导出的剂量-反应关系模型,综合考虑GI和GII型诺如病毒对人群致病力的差异,以及人群易感性及感染后的致病性,将剂量-反应关系模型考虑了四类人群,分别是Se+/GI、Se-/GI、Se+/GII和Se-/GII,其中Se为人组织血型受体(histo-blood group secretor status, Se)。通过生食牡蛎发生诺如病毒食物中毒的剂量-反应关系模型可分为两个阶段,分别是感染模型和感染后发病的模型,其中感染模型的公式如(3)所示: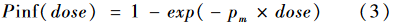 其中pm是贝塔分布输出结果,而dose是通过生食牡蛎摄入的诺如病毒粒子数的泊松分布结果,exp表示为指数函数。估计诺如病毒感染后的发病风险的公式如(4)所示:
其中pm是贝塔分布输出结果,而dose是通过生食牡蛎摄入的诺如病毒粒子数的泊松分布结果,exp表示为指数函数。估计诺如病毒感染后的发病风险的公式如(4)所示: 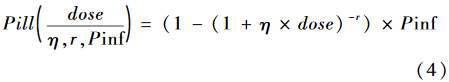 其中η和r是估计感染后发病概率公式的关键参数;四类人群的剂量-反应关系模型参数详见文献[12]。
其中η和r是估计感染后发病概率公式的关键参数;四类人群的剂量-反应关系模型参数详见文献[12]。
2结果与分析
2.1零售阶段牡蛎中诺如病毒的污染水平
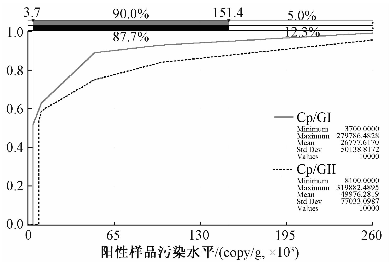
根据对零售市场上采集的牡蛎样品中诺如病毒的检出率和阳性样品的定量检出结果,拟合得到零售阶段牡蛎中GI和GII型诺如病毒的污染水平,拟合结果见图1。其中GI型诺如病毒阳性样品的污染水平均值为2.62×104个病毒拷贝/g[95%置信区间(95%CI):3.73×103~1.54×105],GII型诺如病毒阳性样品的污染水平均值为5.02×104个病毒拷贝/g(95%CI:8.13×103~2.52×105)。
|
图1牡蛎中GI型和GII型诺如病毒的阳性 污染水平拟合结果 Figure 1Fitted cumulative contamination density (copy/gram) for type GI and GII Norovirus in oyster |
2.2居民通过牡蛎可能摄入诺如病毒的量
2.2.1GI型诺如病毒的摄入量
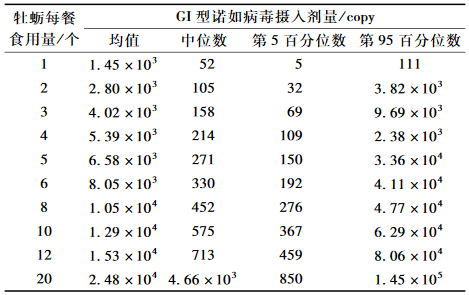
根据评估模型推算,居民在生食情形下食入1、2、…、20只牡蛎可能摄入GI型诺如病毒的量平均值分别为1.45×103,2.80×103,…,2.48×104个基因拷贝。消费不同数量牡蛎可能摄入GI型诺如病毒的剂量分布具体见表2。
|
表2每餐消费不同数量牡蛎可能摄入GI型诺如 病毒的剂量分布 Table 2Estimated distribution of type GI Norovirus intake after consuming various number of oysters |
2.2.2GII型诺如病毒的摄入量
根据评估模型推算,居民在生食情形下食入1、2、…、20只牡蛎可能摄入GII型诺如病毒的量平均值分别为5.16×103、1.06×104、…、1.04×105个基因拷贝。消费不同数量牡蛎可能摄入GII型诺如病毒的剂量分布具体见表3。
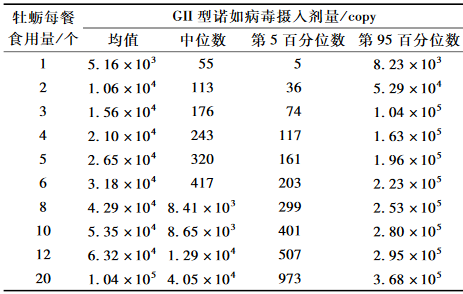
|
表3每餐消费不同数量牡蛎可能摄入GII型诺如 病毒的剂量分布 Table 3Estimated distribution of type GII Norovirus intake after consuming various number of oysters |
2.3风险特征描述
根据上述暴露剂量,结合剂量-反应关系模型,可以得到Se+人群和Se-人群每餐食用不同个数的牡蛎感染GI型诺如病毒和GII型诺如病毒的风险,估计的风险值见详见表4。由此结果可见,对Se+的人群,生食1个可能污染GI和GII型诺如病毒牡蛎的发病风险分别为0.93(95%CI:0.73~0.98)和0.95(95%CI:0.80~0.99);对Se-的人群,生食1个可能污染GI和GII型诺如病毒牡蛎的发病风险分别为0.37(95%CI:0.04~0.64)和0.57(95%CI:0.07~0.99)。对于Se+人群,无论生食被GI型还是GII型诺如病毒污染的牡蛎,消费1~2个牡蛎的平均发病风险就接近1,风险很高;对于Se-人群,生食1个牡蛎的平均发病风险在0.5左右,风险较低,但食用3~4个牡蛎后其发病风险也接近于1,风险很高。

|
表4Se+/Se-人群组对于GI/GII型诺如病毒发病风险的变异性分析结果 Table 4Variability analysis of risks of GI/GII type Norovirus infections for Se+/Se-subpopulations |
2.4敏感性分析
敏感性分析结果显示,发病风险的估计值主要与阴性样品的赋值和污染的可能性相关(图2),尤其是阴性样品的取值,其相关系数为0.49。
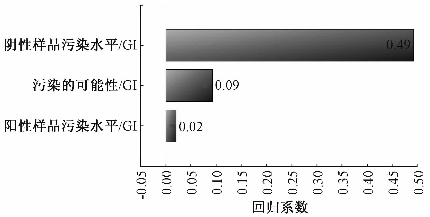
|
图2每餐发病风险的敏感性分析图(以Se+/GI型 每餐一个牡蛎为例) Figure 2Sensitivity analysis of type GI Norovirus infections after consuming one oyster for Se+ populations |
3讨论
在全世界范围内,诺如病毒是引起非细菌性急性感染性胃肠炎的首要病因,约18%(95%CI:17%~20%)的急性胃肠炎的发病与诺如病毒存在关联,在全球食源性诺如病毒暴发的比例约占全部诺如病毒暴发的14%[13]。诺如病毒分为6个基因群,即GI型到GV型,其中GI型和GII型是引起人类急性胃肠炎的主要基因群[14],在被污染的牡蛎中,检出的诺如病毒主要为GI型和GII型[15-17]。由于食品中病毒污染水平的定量数据缺乏、诺如病毒不能在体外进行培养等限制,诺如病毒与食品组合开展风险评估对国内外大多数评估者来说仍是一个挑战,但通过建立传播模式和暴露途径的概念模型,开展暴露评估可以有助于更深入了解病毒的传播路径[18]。随着食品中诺如病毒检测技术和能力的提高,使得定量数据的获得成为可能,但目前牡蛎中诺如病毒污染对人群健康的影响仍缺乏系统性的研究和评估。本研究构建了北京市居民通过生食零售牡蛎可能发生诺如病毒中毒的暴露评估模型,估计了不同人群的发病风险,为进一步评估所需提供的数据支持、相关研究等内容提出建议。由于诺如病毒不能在体外进行培养,对于病毒在环境中沿食品链传递、转移等行为尚未有相关模型,特别是对于不同时间、温度等条件下烹调牡蛎可以导致的病毒的灭活模型尚未见相关报道,因此本评估中很难考虑经烹调后牡蛎中诺如病毒的存活量情况。我国居民牡蛎的消费普遍以白灼、炭烤等方式为主,但为了保留牡蛎的口感,这些烹调方式下中心温度可能只有60~70 ℃。该温度对牡蛎中病毒的灭活效力,尚无详实的研究报道,因此,在基于本次监测数据开展暴露评估时,模拟的是北京市居民生食牡蛎的情景下可能感染诺如病毒的风险。
根据THEBAULT等[12]的剂量-反应关系模型推算,Se+/GI和Se+/GII的半数感染剂量的中位数仅为7.1和1.6个病毒拷贝,而感染后致病的半数剂量的中位数仅为32和4.86个病毒拷贝。尽管本研究对牡蛎中诺如病毒的定量检测取得了较大的进展,但是检出限为每克消化腺100个基因拷贝,远远大于病毒的感染和致病剂量。由于目前对低于检出限的牡蛎中诺如病毒的污染密度未知,本次评估对阴性样品的取值用了均一分布,在0~100之间随机取值,其均值约在50个病毒拷贝,严重高估了人群的发病风险。根据敏感性分析结果可见,阴性样品的取值可极大影响最终发病风险的估计的准确性,这可能由于诺如病毒的感染和发病剂量极低,目前较高的病毒检出限难以定量估计最终的发病风险,因此迫切需要更为精确地病毒拷贝定量检测技术,获得低污染密度下的牡蛎污染情况,从而获得更为可靠的评估数据。研究结果提示,基于现有检测技术,生食诺如病毒检出阳性的牡蛎发生诺如病毒感染的风险非常高,建议居民尽可能避免生食牡蛎。收集诺如病毒随温度发生变化的相关数据,建立随牡蛎烹调温度变化而改变的诺如病毒动态模型,提高牡蛎中诺如病毒定量检测的敏感性,是进一步开展牡蛎中诺如病毒定量风险评估的重要数据支持和依据。
参考文献
[1]World Health Organization. WHO estimates of the global burden of foodborne diseases: foodborne disease burden epidemiology reference group 2007-2015[R]. Geneva: WHO, 2015:1-268.
[2]SCALLAN E, HOEKSTRA R M, ANGULO F J, et al. Foodborne illness acquired in the United States-major pathogens[J]. Emerg Infect Dis,2011, 17(1): 7-15. DOI: 10.3201/eid1701.P11101.
[3]王晓欢,于恩庶. 诺如病毒胃肠炎的研究进展[J]. 中国人兽共患病学报, 2007,23(6):621-624.
[4]MESQUITA J R, NASCIMENTO M S. A foodborne outbreak of Norovirus gastroenteritis associated with a Christmas dinner in Porto, Portugal, December 2008[J]. Euro Surveill, 2009,14(41):19355.
[5]ISAKBAEVA E T, WIDDOWSON M A, BEARD R S, et al. Norovirus transmission on cruise ship [J]. Emerg Infect Dis, 2005,11(1):154-158.
[6]BURKHARDT W R, CALCI K R. Selective accumulation may account for shellfish-associated viral illness [J]. Appl Environ Microbiol, 2000,66(4):1375-1378.
[7]SHIEH Y C, MONROE S S, FANKHAUSER R L, et al. Detection of Norwalk-like virus in shellfish implicated in illness[J]. Journal of Infectious Diseases, 2000,181(Supplement 2):S360-S366.
[8]WESTRELL T, DUSCH V, ETHELBERG S, et al. Norovirus outbreaks linked to oyster consumption in the United Kingdom, Norway, France, Sweden and Denmark, 2010[J]. Euro Surveill, 2010,15(12):321-334.
[9]MCINTYRE L, GALANIS E, MATTISON K, et al. Multiple clusters of Norovirus among shellfish consumers linked to symptomatic oyster harvesters[J]. J Food Prot, 2012,75(9):1715-1720.
[10]江涛,韩春卉,张宏元,等. 北京市市售牡蛎中诺如病毒核酸检测及定量分析[J]. 中国食品卫生杂志,2017,29(2):126-130.
[11]VOSE D. Risk analysis: a quantitative guide,3rd edition[M]. New York: Wiley, 2000:599-688.
[12]THEBAULT A, TEUNIS P F M, LE PENDU J, et al. Infectivity of GI and GII Noroviruses established from oyster related outbreaks [J]. Epidemics, 2013,5(2): 98-101.
[13]宋夏,刘弘.诺如病毒的流行病学研究 [J].上海预防医学,2017,29(4):261-272.
[14]VINJE J. Advances in laboratory methods for detection and typing of Norovirus [J]. J Clin Microbiol, 2015, 53(2):373-381.
[15]LOWTHER J A, GUSTAR N E, POWELL A L, et al. Two-year systematic study to assess Norovirus contamination in oysters from commercial harvesting areas in the United Kingdom [J]. Appl Environ Microbiol, 2012,78(16):5812-5817.
[16]NISHIDA T, KIMURA H, SAITOH M, et al. Detection, quantitation, and phylogenetic analysis of Noroviruses in Japanese oysters [J]. Appl Environ Microbiol, 2003,69(10):5782-5786.
[17]柯丹枫, 张永慧. 广东养殖牡蛎的诺如病毒污染状况调查[J]. 中华疾病控制杂志, 2012,16(10):881-884.
[18]FAO/WHO.Virus in food: scientific advice to support risk management activities[R]. Microbiological Risk Assessment Series No. 13, 2008.
[2]SCALLAN E, HOEKSTRA R M, ANGULO F J, et al. Foodborne illness acquired in the United States-major pathogens[J]. Emerg Infect Dis,2011, 17(1): 7-15. DOI: 10.3201/eid1701.P11101.
[3]王晓欢,于恩庶. 诺如病毒胃肠炎的研究进展[J]. 中国人兽共患病学报, 2007,23(6):621-624.
[4]MESQUITA J R, NASCIMENTO M S. A foodborne outbreak of Norovirus gastroenteritis associated with a Christmas dinner in Porto, Portugal, December 2008[J]. Euro Surveill, 2009,14(41):19355.
[5]ISAKBAEVA E T, WIDDOWSON M A, BEARD R S, et al. Norovirus transmission on cruise ship [J]. Emerg Infect Dis, 2005,11(1):154-158.
[6]BURKHARDT W R, CALCI K R. Selective accumulation may account for shellfish-associated viral illness [J]. Appl Environ Microbiol, 2000,66(4):1375-1378.
[7]SHIEH Y C, MONROE S S, FANKHAUSER R L, et al. Detection of Norwalk-like virus in shellfish implicated in illness[J]. Journal of Infectious Diseases, 2000,181(Supplement 2):S360-S366.
[8]WESTRELL T, DUSCH V, ETHELBERG S, et al. Norovirus outbreaks linked to oyster consumption in the United Kingdom, Norway, France, Sweden and Denmark, 2010[J]. Euro Surveill, 2010,15(12):321-334.
[9]MCINTYRE L, GALANIS E, MATTISON K, et al. Multiple clusters of Norovirus among shellfish consumers linked to symptomatic oyster harvesters[J]. J Food Prot, 2012,75(9):1715-1720.
[10]江涛,韩春卉,张宏元,等. 北京市市售牡蛎中诺如病毒核酸检测及定量分析[J]. 中国食品卫生杂志,2017,29(2):126-130.
[11]VOSE D. Risk analysis: a quantitative guide,3rd edition[M]. New York: Wiley, 2000:599-688.
[12]THEBAULT A, TEUNIS P F M, LE PENDU J, et al. Infectivity of GI and GII Noroviruses established from oyster related outbreaks [J]. Epidemics, 2013,5(2): 98-101.
[13]宋夏,刘弘.诺如病毒的流行病学研究 [J].上海预防医学,2017,29(4):261-272.
[14]VINJE J. Advances in laboratory methods for detection and typing of Norovirus [J]. J Clin Microbiol, 2015, 53(2):373-381.
[15]LOWTHER J A, GUSTAR N E, POWELL A L, et al. Two-year systematic study to assess Norovirus contamination in oysters from commercial harvesting areas in the United Kingdom [J]. Appl Environ Microbiol, 2012,78(16):5812-5817.
[16]NISHIDA T, KIMURA H, SAITOH M, et al. Detection, quantitation, and phylogenetic analysis of Noroviruses in Japanese oysters [J]. Appl Environ Microbiol, 2003,69(10):5782-5786.
[17]柯丹枫, 张永慧. 广东养殖牡蛎的诺如病毒污染状况调查[J]. 中华疾病控制杂志, 2012,16(10):881-884.
[18]FAO/WHO.Virus in food: scientific advice to support risk management activities[R]. Microbiological Risk Assessment Series No. 13, 2008.
 宋筱瑜,李凤琴,江涛,韩春卉,李楠,王佳慧,徐海滨,朱江辉.北京市市售牡蛎中诺如病毒污染对居民健康影响的初步定量风险评估[J].中国食品卫生杂志,2018,30(1):79-83.
宋筱瑜,李凤琴,江涛,韩春卉,李楠,王佳慧,徐海滨,朱江辉.北京市市售牡蛎中诺如病毒污染对居民健康影响的初步定量风险评估[J].中国食品卫生杂志,2018,30(1):79-83.