DOi:10.13590/j.cjfh.2018.02.001
基质辅助激光解吸电离飞行时间质谱技术对阪崎克罗诺杆菌的鉴定及溯源
(1.大连工业大学生物工程学院,辽宁 大连116034; 2.中国检验检疫科学研究院, 北京100176; 3.国家乳制品质量监督检验中心,黑龙江 哈尔滨150086)
收稿日期:2018-03-01
作者简介:王伟女硕士生研究方向为微生物溯源方法E-mail: 3169324291@qq.com
通信作者:卢行安男研究员研究方向为微生物检验质量控制和溯源方法E-mail: luxa@sina.com
基金项目:科技部“食品安全关键技术研发”重点专项(2017YFC1601401)
摘要:目的研究不同样品前处理方法对VITEK基质辅助激光解吸电离飞行时间质谱(VITEK MALDI-TOF MS,以下简称VITEK MS)鉴定阪崎克罗诺杆菌的影响及该方法对阪崎克罗诺杆菌的分型溯源能力,研究VITEK MS与MALDI 7090TM MALDI-TOF/TOF MS(以下简称MALDI 7090)所得图谱的异同。方法选取3种不同样品前处理方法处理18株阪崎克罗诺杆菌野生菌株、2株阪崎克罗诺杆菌标准菌株和2株非阪崎克罗诺杆菌标准菌株,比较VITEK MS鉴定结果与传统鉴定结果的一致性;通过VITEK MS的鉴定结果和图谱的比对,研究不同样品前处理方法对阪崎克罗诺杆菌鉴定的影响;利用VITEK MS质谱图特征峰的聚类分析,对20株阪崎克罗诺杆菌进行溯源研究;同时比较VITEK MS与MALDI 7090所得图谱,研究两者的异同。结果20株试验菌株的VITEK MS鉴定结果均为阪崎克罗诺杆菌,与传统的鉴定结果一致,并与2株非阪崎克罗诺杆菌标准菌株能明显地区分;3种不同样品前处理方法得到的鉴定谱图与标准谱图比对获得的鉴定结果可信度之间差异无统计学意义(P>0.05),但质谱图中质谱峰数量和相对峰强度有明显差异,其中甲酸提取法在2 000~15 000 m/z范围内,相对峰强度>5 mV的质谱峰数量达15个以上。在相似水平为80%的条件下,VITEK MS的聚类分析将甲酸提取法的20株阪崎克罗诺杆菌分为5个群,通过聚类分析及菌株来源能够推测阪崎克罗诺杆菌传播途径。VITEK MS和MALDI 7090得到的谱图,两者特征峰的出峰位置与相对峰强度无明显差异。结论MALDI-TOF MS技术可以准确鉴定阪崎克罗诺杆菌;甲酸提取法更适用于阪崎克罗诺杆菌的MALDI-TOF MS鉴定;MALDI-TOF MS技术对阪崎克罗诺杆菌的溯源研究具有重要价值;VITEK MS与MALDI 7090所得谱图无明显差异。
关键词:
阪崎克罗诺杆菌; 基质辅助激光解吸电离飞行时间质谱; 鉴定; 样品前处理; 聚类分析; 溯源; 食源性致病菌
文章编号:1004-8456(2018)02-0125-07
中图分类号:R155
文献标志码:A
The identification of Cronobacter sakazakii and its traceability by matrix-assisted laser desorption ionization-time of flight mass spectrometry
(1.School of Biological Engineering, Dalian Polytechnic University, Liaoning Dalian 116034, China; 2.Chinese Academy of Inspection and Quarantine, Beijing 100176,China; 3.National Dairy Engineering and Technical Research Center, Heilongjiang Ha)
Abstract:Objective VITEK matrix-assisted laster desorption ionization-time of flight mass spectrometry (VITEK MALDI-TOF MS,hereinafter referred to as VITEK MS). To study the impact of different sample preparation procedures for Cronobacter sakazakii identification and its traceability. And two data acquisition softwares of VITEK MS and MALDI 7090 were used to compare the differences of mass spectrograms between them. Methods Twenty two strains, including 18 isolated Cronobacter sakazakii strains, two Cronobacter sakazakii reference strains, one Enterobacter cloacae reference strain and one Citrobacter freundii reference strain were prepared respectively by three different preparation procedures, including direct transfer method, formic acid extraction method and formic acid-acetonitrile extraction method, and analyzed by MS. A detailed analysis of mass spectrograms was obtained from the software of VITEK MS. The result were compared with biochemical result. Then, the impact of sample preparation method on the identification result was evaluated by researching the consistency of result derived from three different preparation method, The traceability of 20 Cronobacter sakazakii strains was also investigated by the cluster analysis of VITEK MS result. The same strains were analyzed by MALDI 7090, and the mass spectrograms were compared with those by VITEK MS. ResultsThe 20 strains were identified by VITEK MS as Cronobacter sakazakii, which were in consistent with biochemical result and could be distinguished from Enterobacter cloacae and Citrobacter freundii. There was no significant difference in the result confidence among the three different preparation procedures(P>0.05), but the number of mass peaks and their relative intensities showed remarkable difference. The formic acid extraction method achieved spectra consisting of >15 peaks and mass ranges of 2 000-15 000 m/z, of which relative intensities were >5 mV. At similarity level of 80%, the 20 strains prepared by the formic acid extraction method were classified into 5 clusters by the cluster analysis of VITEK MS result, through which the transmission route of Cronobacter sakazakii could be identified. There was no obvious difference in the peak positions and relative intensity between VITEK MS and MALDI 7090. ConclusionMALDI-TOF MS was considered as a reliable technique in the identification of Cronobacter sakazakii. The formic acid extraction method was more suitable for the identification of Cronobacter sakazakii by MALDI-TOF MS, which will be a useful technique for Cronobacter sakazakii trace. There was no obvious difference in the mass spectrograms between VITEK MS and MALDI 7090.
Key words:
Cronobacter sakazakii; matrix-assisted laser desorption ionization-time of flight mass spectrometry; identification; sample preparation procedure; cluster analysis; traceability; foodborne pathogenic bacteria
克罗诺杆菌属是一种革兰阴性菌的条件致病菌,会引起严重威胁人类生命的疾病[1]。早在1961年,URMENYI和FRANKLIN[2]发表了有关感染克罗诺杆菌属的报道,当时克罗诺杆菌属被命名为黄色阴沟杆菌(yellow-pigmented Enterobacter cloacae),直到1980年被更名为阪崎肠杆菌(Enterobacter sakazakii)[3-4]。2008年IVERSEN等[5]通过试验将阪崎肠杆菌准确的定义为克罗诺杆菌属,属于肠杆菌科。阪崎克罗诺杆菌是克罗诺杆菌属的一个种,会引起新生儿坏死性小肠结肠炎、脑膜炎、败血症等疾病[6-7],易感人群是新生儿、婴儿及老年人[4],可引起神经系统后遗症和死亡,死亡率达40%~80%[3]。传统的检测方法存在检测周期长、操作繁琐等缺陷,因此,建立一种高效、简便的阪崎克罗诺杆菌鉴定方法对于相关疾病的调查、溯源和阻断其传播途径具有重要意义。
基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)是一种新型软电离技术,其基本工作原理是待测微生物与基质形成共结晶体,基质吸收激光能量,并将能量及H+(质子)传递给微生物所含生物大分子(主要是核糖体蛋白质),使其发生电离;在电场作用下,带电荷离子离开共结晶体表面,进入一定长度的真空管,其到达真空管顶端的离子检测器的时间与其质量成反比,从而获得蛋白质指纹图谱,并与数据库中的标准谱图进行比对分析,从而获得鉴定结果。在蛋白质指纹图谱中,横坐标表示离子的质荷比(m/z),纵坐标表示离子峰强度,因此可利用特征峰值、特征峰数量及相对峰强度对图谱进行比较[8]。在我国MALDI-TOF MS技术检测微生物才刚刚兴起,一些影响鉴定结果的因素还处在摸索阶段,例如样品前处理方法、不同的仪器等[9]。本研究应用VITEK MALDI-TOF MS(以下简称VITEK MS)对20株阪崎克罗诺杆菌进行鉴定,研究不同样品前处理方法对VITEK MS鉴定阪崎克罗诺杆菌的影响及VITEK MS对阪崎克罗诺杆菌的分型溯源能力,并通过VITEK MS与MALDI 7090TM MALDI-TOF/TOF MS(以下简称MALDI 7090)的图谱比对,研究两者所得谱图的异同。
哥伦比亚血琼脂+5%绵羊血、2-氰基-4-羟基肉桂酸基质均购自法国梅里埃,无菌水,无水乙醇、甲酸、乙腈均为色谱纯。
羊血)平板上,置于36 ℃培养24 h,使细菌形成1 mm大小的菌落备用。
甲酸提取法(Ⅱ法):1.5 ml Eppendorf管中加入1 ml 70%乙醇,用无菌棉棒挑取血平板上的单个菌落(约5 mg)放入70%乙醇中清洗,15 000 r/min离心5 min,去掉上清液,然后加入100 μl 25%甲酸15 000 r/min离心5 min,将上清液转移到新的Eppendorf管中备用[10]。取1 μl上述备用的上清液滴加在质谱仪靶板的靶点上,再加入1 μl 2-氰基-4-羟基肉桂酸基质液均匀覆盖,待自然晾干后即可进行检测。
甲酸-乙腈提取法(Ⅲ法):1.5 ml Eppendorf管中加入300 μl纯水,用无菌棉棒挑取血平板上的单个菌落(约5 mg)放入纯水中清洗,然后加入900 μl无水乙醇,充分混匀,12 000 r/min离心2 min,弃去上清液,向含有沉淀的Eppendorf管中加入50 μl 70%的甲酸,混匀后,再加入50 μl乙腈,充分混匀后,12 000 r/min离心2 min,将上清液转移到新的Eppendorf管中备用。取1 μl上述备用的上清液滴加在质谱仪靶板的靶点上,再加入1 μl 2-氰基-4-羟基肉桂酸基质液均匀覆盖,待自然晾干后即可进行检测。
仪器参数:氮气激光光源,激光频率50~60 Hz,激光波长337 nm,加速电压20 000 V,延迟提取电压2 100 V,飞行管长度1.2 m,线性正离子采样模式,设定质量范围为2 000~20 000 Da。每次检测待测样品的同时都要在质控靶点的位置用大肠埃希菌标准菌株(ATCC 8739)进行质量校正,以降低结果数据的误差。
软件分析:利用采集软件Launch Pad进行数据采集,获取质谱图。激光采样点击靶点5次,可获取1个质谱图,每个靶位取多个位置进行激光点击,获得100个有效的质谱图汇总并与数据库中的标准图谱进行匹配,由SARAMIS软件自动给出鉴定结果。其中,鉴定结果可信度用百分比(%)表示,并在SARAMIS结果窗口用不同的颜色标识,深绿色表示可信度大于99.9%,绿色表示可信度在90.0%~99.8%之间,黄色表示可信度在80.0%~89.9%之间,白色表示可信度在75.0%~79.9%之间,红色表示样品的质谱图与数据库中两个或两个以上标准谱图匹配的可信度大于80.0%,为“冲突结果”,当可信度小于75.0%,结果窗口不显示,在Details里给出鉴定结果和可信度。
仪器参数:固态紫外线激光光源,最大激光重复频率2 000 Hz,激光波长355 nm,加速电压20 000 V,延迟提取电压2 000~2 400 V,飞行管长度2.1 m,线性正离子采样模式,设定质量范围为2 000~20 000 Da。每次检测待测样品的同时都要用大肠埃希菌标准菌株(ATCC 8739)进行质量校正,以降低结果数据的误差。
软件分析:利用软件MALDI Solutions进行数据采集,获取质谱图。激光采样点击靶点100次,可获取1个质谱图,每个靶位取多个位置进行激光点击,获得100个有效的质谱图,经平均与峰型处理,获取最终的质谱图。
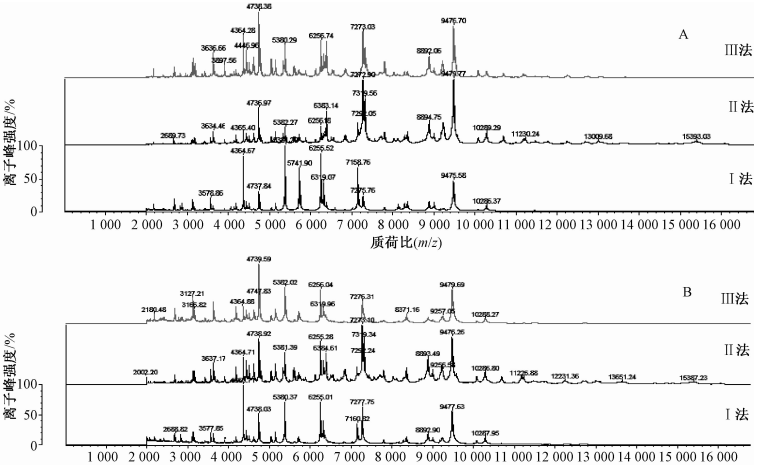
Ⅱ法的3株标准菌株(ATCC 51329、ATCC 13047、ATCC 8090)得到的质谱谱图见图2。从图中可以看出3株标准菌株的质谱峰差异明显,ATCC 51329的质谱峰4 736、6 383、7 272、7 319、9 479 m/z是其他2株标准菌株没有的,7 272 m/z的相对峰强度最高;ATCC 13047的质谱峰1 998、3 624、4 755、7 247、9 509 m/z是其他2株标准菌株没有的,7 247 m/z的相对峰强度最高;ATCC 8090的质谱峰2 181、4 762、5 240、7 261、7 334、9 555 m/z是其他2株标准菌株没有的,4 762 m/z的相对峰强度最高。说明MALDI-TOF MS能准确区分不同种属的菌株。
通过20株阪崎克罗诺杆菌的谱图比对(图略,举例如图1),3种不同样品前处理方法得到的谱图在4 364、4 739、5 381、6 255、7 272和9 477 m/z附近均有质谱峰出现,Ⅰ法大部分菌株在4 364 m/z的相对峰强度最高,Ⅱ法大部分菌株在7 272 m/z的相对峰强度最高,Ⅲ法大部分菌株在4 739 m/z的相对峰强度最高,相对峰强度存在差异,这些质谱峰可作为阪崎克诺杆菌的特征峰。
通过对这20株阪崎克罗诺杆菌的质谱图进行比较,Ⅰ群的IE11、IE12和Ⅱ群、Ⅲ群、Ⅳ群的19、IE7、ATCC 29544的质谱峰出现差异。IE11和IE12共有的质谱峰6 302、9 579 m/z是其他菌株没有的;19的质谱峰8 373 m/z是其他菌株没有的,并且在4 363 m/z左右相对峰强度最高;IE7的质谱峰7 343 m/z是其他菌株没有的;ATCC 29544的质谱峰3 579、7 158 m/z是其他菌株没有的。优势群Ⅴ的菌株大多数在相同的位置有不同强度的质谱峰出现。说明MALDI-TOF MS具有良好的聚类分型能力。
理方法获取的质谱峰数量不同,相同的质谱峰对应的相对峰强度也不同,表明3种不同样品前处理方法提纯的蛋白质出现差异,甲酸提取法获得相对峰强度>5 mV的质谱峰数量较多,并且分布范围更宽,因此,甲酸提取法较适用于阪崎克罗诺杆菌的MALDI-TOF MS鉴定。赵贵明等[11]选取直接涂布法和甲酸-乙腈提取法处理1株阪崎克罗诺杆菌标准菌株,来确定MALDI-TOF MS鉴定的最佳样品前处理方法,得出两种方法的谱图中蛋白质离子峰的强度一致,与本研究中的结论不同,可能是由于其研究菌株数量较少所致。李修远等[12]列举了4种样品前处理方法对不同微生物应用MALDI-TOF MS鉴定的影响,传统直涂法用于革兰阴性杆菌的效果最佳,改良直涂法和甲酸-乙腈提取法用于革兰阴性杆菌、革兰阴性球菌和革兰阳性球菌的效果最佳,研磨-超声裂解法几乎适用于所有的微生物,这些研究表明为获得更好鉴定结果,不同种类细菌需要不同处理方法。本研究选取了一种新的样品前处理方法,即甲酸提取法,结果表明其更适用于阪崎克罗诺杆菌的MALDI-TOF MS鉴定,对于其他菌种的鉴定效果还有待考察。由 于菌株自身的特征(荚膜、细胞壁厚度等的差异)[13],还有菌落表面粘连的代谢物、色素、琼脂等杂质,样品前处理方法直接影响蛋白质的提纯,是影响MALDI-TOF MS鉴定结果的重要因素,而不同的微生物最佳样品前处理方法也不同;因此,样品前处理方法对MALDI-TOF MS鉴定微生物的影响还需做进一步的研究。
通过VITEK MS对甲酸提取法的20株阪崎克罗诺杆菌的聚类分析得出,在相关性为80%的条件下,20株阪崎克罗诺杆菌分为5个群,说明MALDI-TOF MS技术能区分同种菌株间的微小差异,根据菌株来源,推测优势群Ⅴ中14株野生菌株传播途径为草→进风口→取风室→采样人员→实验室→排风口,表明MALDI-TOF MS技术对阪崎克罗诺杆菌分型溯源具有重要价值。吕佳等[14]利用MALDI-TOF MS技术对4株肠炎沙门菌进行聚类分析,并将其结果与主成分分析结果进行比较,得出MALDI-TOF MS技术可为沙门菌分型溯源提供一种新的手段,由于本研究中试验菌株的数量有限,因此在这方面还需做进一步的探究。
VITEK MS与MALDI 7090的质谱图比对的结果表明,两者特征峰出峰位置与相对峰强度无太大差异,证明了基质辅助激光解吸的可靠性,由于MALDI 7090没有微生物数据库,并且获得的图谱无法转换为txt文件,因此只能比较两者谱图的异同,两者对微生物鉴定结果的异同还有待考察。
综上,MALDI-TOF MS技术应用于微生物的鉴定及溯源具有快速、简便和准确的优点,但也存在一些影响鉴定结果准确性和重复性的因素,另外,数据库中菌株信息的不全面和具有高效分辨力软件的缺乏,也是目前MALDI-TOF MS技术面临的挑战[15];因此,最大限度的发挥MALDI-TOF MS技术的优势,确保鉴定结果的准确性和可靠性,对于食源性致病菌的检测与鉴定、分型溯源等都具有重大意义。
基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)是一种新型软电离技术,其基本工作原理是待测微生物与基质形成共结晶体,基质吸收激光能量,并将能量及H+(质子)传递给微生物所含生物大分子(主要是核糖体蛋白质),使其发生电离;在电场作用下,带电荷离子离开共结晶体表面,进入一定长度的真空管,其到达真空管顶端的离子检测器的时间与其质量成反比,从而获得蛋白质指纹图谱,并与数据库中的标准谱图进行比对分析,从而获得鉴定结果。在蛋白质指纹图谱中,横坐标表示离子的质荷比(m/z),纵坐标表示离子峰强度,因此可利用特征峰值、特征峰数量及相对峰强度对图谱进行比较[8]。在我国MALDI-TOF MS技术检测微生物才刚刚兴起,一些影响鉴定结果的因素还处在摸索阶段,例如样品前处理方法、不同的仪器等[9]。本研究应用VITEK MALDI-TOF MS(以下简称VITEK MS)对20株阪崎克罗诺杆菌进行鉴定,研究不同样品前处理方法对VITEK MS鉴定阪崎克罗诺杆菌的影响及VITEK MS对阪崎克罗诺杆菌的分型溯源能力,并通过VITEK MS与MALDI 7090TM MALDI-TOF/TOF MS(以下简称MALDI 7090)的图谱比对,研究两者所得谱图的异同。
1材料与方法
1.1材料
1.1.1试验菌株
从乳制品实验室和外界环境中分离的18株阪崎克罗诺杆菌野生菌株,其结果均由传统生化鉴定得出,2株阪崎克罗诺杆菌标准菌株(ATCC 51329、ATCC 29544),1株阴沟肠杆菌(Enterobacter cloacae)标准菌株(ATCC 13047),1株弗氏枸橼酸杆菌(Citrobacter freundii)标准菌株(ATCC 8090),共22株菌株,试验菌株信息见表1。
1.1.2主要仪器与试剂
VITEK MALDI-TOF MS(法国生物梅里埃),MALDI 7090 TM MALDI-TOF/TOF Mass Spectrometer(日本岛津)。哥伦比亚血琼脂+5%绵羊血、2-氰基-4-羟基肉桂酸基质均购自法国梅里埃,无菌水,无水乙醇、甲酸、乙腈均为色谱纯。
1.2方法
1.2.1菌株活化
将试验菌株接种于哥伦比亚血琼脂(含5%绵
|
表1试验菌株信息 Table 1 Basic information of Cronobacter sakazakii strains used in this study |
1.2.2样品前处理
直接涂布法(Ⅰ法):用1 μl接种环从血平板上挑取单个待测菌落,涂布到靶板上,然后加入1 μl 2-氰基-4-羟基肉桂酸基质液均匀覆盖,待自然晾干后即可进行检测。甲酸提取法(Ⅱ法):1.5 ml Eppendorf管中加入1 ml 70%乙醇,用无菌棉棒挑取血平板上的单个菌落(约5 mg)放入70%乙醇中清洗,15 000 r/min离心5 min,去掉上清液,然后加入100 μl 25%甲酸15 000 r/min离心5 min,将上清液转移到新的Eppendorf管中备用[10]。取1 μl上述备用的上清液滴加在质谱仪靶板的靶点上,再加入1 μl 2-氰基-4-羟基肉桂酸基质液均匀覆盖,待自然晾干后即可进行检测。
甲酸-乙腈提取法(Ⅲ法):1.5 ml Eppendorf管中加入300 μl纯水,用无菌棉棒挑取血平板上的单个菌落(约5 mg)放入纯水中清洗,然后加入900 μl无水乙醇,充分混匀,12 000 r/min离心2 min,弃去上清液,向含有沉淀的Eppendorf管中加入50 μl 70%的甲酸,混匀后,再加入50 μl乙腈,充分混匀后,12 000 r/min离心2 min,将上清液转移到新的Eppendorf管中备用。取1 μl上述备用的上清液滴加在质谱仪靶板的靶点上,再加入1 μl 2-氰基-4-羟基肉桂酸基质液均匀覆盖,待自然晾干后即可进行检测。
1.2.3MALDI-TOF MS分析
1.2.3.1VITEK MS分析
上样:使用4×48孔靶板,将处理后的样品点至靶板,并覆盖基质溶液,室温自然晾干后上机分析。仪器参数:氮气激光光源,激光频率50~60 Hz,激光波长337 nm,加速电压20 000 V,延迟提取电压2 100 V,飞行管长度1.2 m,线性正离子采样模式,设定质量范围为2 000~20 000 Da。每次检测待测样品的同时都要在质控靶点的位置用大肠埃希菌标准菌株(ATCC 8739)进行质量校正,以降低结果数据的误差。
软件分析:利用采集软件Launch Pad进行数据采集,获取质谱图。激光采样点击靶点5次,可获取1个质谱图,每个靶位取多个位置进行激光点击,获得100个有效的质谱图汇总并与数据库中的标准图谱进行匹配,由SARAMIS软件自动给出鉴定结果。其中,鉴定结果可信度用百分比(%)表示,并在SARAMIS结果窗口用不同的颜色标识,深绿色表示可信度大于99.9%,绿色表示可信度在90.0%~99.8%之间,黄色表示可信度在80.0%~89.9%之间,白色表示可信度在75.0%~79.9%之间,红色表示样品的质谱图与数据库中两个或两个以上标准谱图匹配的可信度大于80.0%,为“冲突结果”,当可信度小于75.0%,结果窗口不显示,在Details里给出鉴定结果和可信度。
1.2.3.2MALDI 7090分析
上样:使用4×48孔靶板,将处理后的样品点至靶板,并覆盖基质溶液,室温自然晾干后上机分析。仪器参数:固态紫外线激光光源,最大激光重复频率2 000 Hz,激光波长355 nm,加速电压20 000 V,延迟提取电压2 000~2 400 V,飞行管长度2.1 m,线性正离子采样模式,设定质量范围为2 000~20 000 Da。每次检测待测样品的同时都要用大肠埃希菌标准菌株(ATCC 8739)进行质量校正,以降低结果数据的误差。
软件分析:利用软件MALDI Solutions进行数据采集,获取质谱图。激光采样点击靶点100次,可获取1个质谱图,每个靶位取多个位置进行激光点击,获得100个有效的质谱图,经平均与峰型处理,获取最终的质谱图。
1.3统计学分析
通过配对资料t检验来评价两种方法检测结果之间的差异,以P<0.05为差异有统计学意义。
2结果与分析
2.1VITEK MS的鉴定结果分析
2.1.1试验菌株的鉴定结果
按照1.2.2中的样品前处理方法处理试验菌株,利用VITEK MS进行鉴定。22株试验菌株的鉴定结果与传统鉴定结果一致,标准菌株ATCC 13047鉴定为阴沟肠杆菌,标准菌株ATCC 8090鉴定为弗氏枸橼酸杆菌,另外20株菌株均鉴定为阪崎克罗诺杆菌,鉴定符合率为100.0%。
2.1.23种不同样品前处理方法的鉴定结果
将3种不同样品前处理方法得到的鉴定谱图与超级谱库中的标准谱图进行比对,得到鉴定结果可信度,对鉴定结果可信度进行汇总,见表2,并对其进行配对资料t检验P值均>0.05,表明3种不同样品前处理方法的鉴定结果可信度之间差异无统计学意义(P>0.05)。|
|
表23种不同样品前处理方法鉴定结果分析(%) Table 2Identification results analysis of three different preparation procedures |
2.1.33种不同样品前处理方法的图谱比较
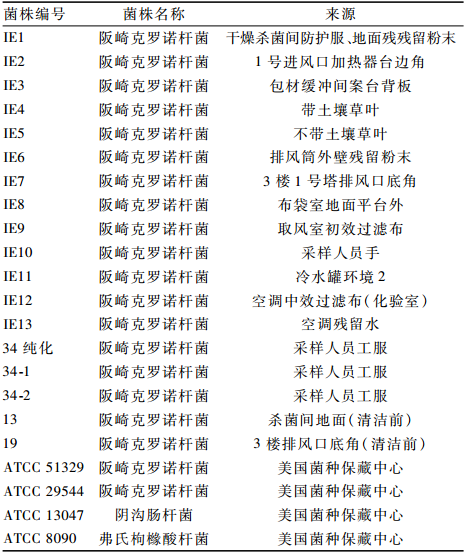
用VITEK MS鉴定试验菌株,由SARAMIS采集样品质谱图,图1为ATCC 51329和IE10的VITEK MS质谱图。从图中可以看出,3种不同样品前处理方法获得的质谱图,质谱峰数量不同,相同的质谱峰对应的相对峰强度也不同,其中Ⅱ法在2 000~15 000 m/z范围内,相对峰强度>5 mV的质谱峰数量达15个以上(如ATCC 51329,见表3);因此,应用MALDI-TOF MS鉴定阪崎克罗诺杆菌,样品前处理方法对鉴定谱图存在一定程度的影响,Ⅱ法较适用于阪崎克罗诺杆菌的MALDI-TOF MS鉴定。注:A为ATCC 51329;B为IE10
|
图1ATCC 51329和IE10的VITEK MS图谱 Figure 1Mass spectrograms of ATCC 51329 and IE10 for VITEK MS analysis |
|
|
表33种不同样品前处理方法的图谱比较 Table 3Mass spectrograms comparation of three different preparation procedures 注:以ATCC 51329为例 |

|
图2Ⅱ法3株标准菌株的VITEK MS质谱图谱 Figure 2Mass spectrograms of three standard strains prepared by method Ⅱfor VITEK MS analysis |
通过20株阪崎克罗诺杆菌的谱图比对(图略,举例如图1),3种不同样品前处理方法得到的谱图在4 364、4 739、5 381、6 255、7 272和9 477 m/z附近均有质谱峰出现,Ⅰ法大部分菌株在4 364 m/z的相对峰强度最高,Ⅱ法大部分菌株在7 272 m/z的相对峰强度最高,Ⅲ法大部分菌株在4 739 m/z的相对峰强度最高,相对峰强度存在差异,这些质谱峰可作为阪崎克诺杆菌的特征峰。
2.2VITEK MS的聚类分析
将Ⅱ法的20株阪崎克罗诺杆菌的质谱图导入VITEK MS系统的数据库中,应用SARAMIS软件对其进行聚类分析,在相关性为80%的条件下,20株阪崎克罗诺杆菌分为5个群,聚类分析图见图3。从聚类分析结果可以看出,IE11和IE12被归为Ⅰ群,19、IE7、ATCC 29544分别被归为Ⅱ群、Ⅲ群、Ⅳ群,说明冷水罐环境2与空调中效过滤布(化验室)可能污染了同一种阪崎克罗诺杆菌,而3楼排风口底角可能污染了不同菌株来源的阪崎克罗诺杆菌;优势群Ⅴ共有15株菌,占比75%(15/20),其中有1株标准菌株ATCC 51329,说明这14株野生菌株与标准菌株ATCC 51329之间亲缘关系非常近,根据14株野生菌株的来源推测其传播途径如下:草→进风口→取风室→采样人员→实验室→排风口。
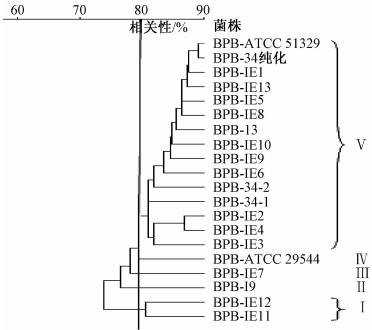
|
注:BPB表示甲酸提取法 图320株阪崎克罗诺杆菌聚类分析图 Figure 3Dendrograms of 20 Cronobacter sakazakii strains |
2.3VITEK MS与MALDI 7090的图谱比较
利用MALDI 7090对上述试验菌株进行分析,由MALDI Solutions采集样品质谱图,将VITEK MS与MALDI 7090的图谱进行比较,图4为ATCC 51329和IE10的MALDI 7090质谱图。Ⅱ法IE10的VITEK MS与MALDI 7090谱图中均存在特征峰4 364、4 739、5 381、6 255、7 272和9 477 m/z,将特征峰进行汇总,见图5,表明两者特征峰出峰位置与相对峰强度无明显差异。
3讨论
VITEK MS的鉴定结果表明,3种不同样品前处理方法获得的鉴定结果可信度,配对资料t检验的P值均>0.05,表明鉴定结果可信度之间差异无统计学意义;但从质谱图中看出,3种不同样品前处
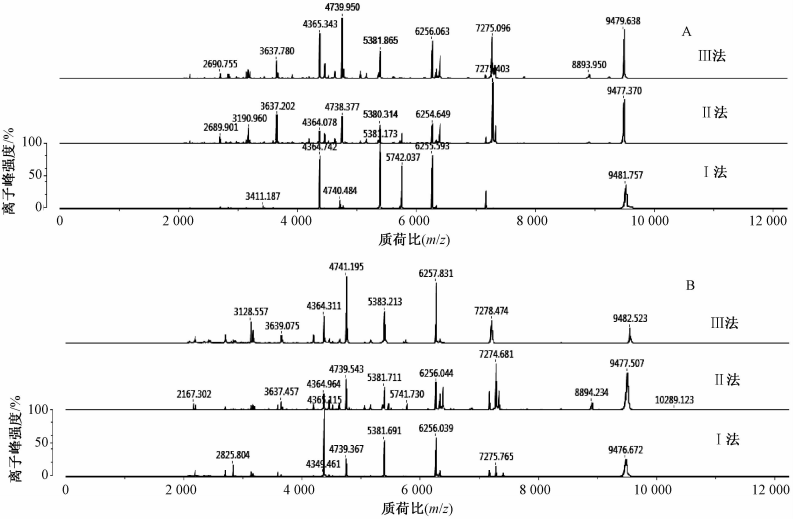
|
注:A为ATCC 51329;B为IE10 图4ATCC 51329和IE10的MALDI 7090图谱 Figure 4Mass spectrograms of ATCC 51329 and IE10 for MALDI 7090 analysis |

|
注:a、b、c、d、e、f分别表示特征峰4 364、4 739、5 381、6 255、7 272 和9 477 m/z 图5Ⅱ法IE10的特征峰强度分布图 Figure 5Characteristic peak intensity distribution map of IE10 prepared by method Ⅱ |
通过VITEK MS对甲酸提取法的20株阪崎克罗诺杆菌的聚类分析得出,在相关性为80%的条件下,20株阪崎克罗诺杆菌分为5个群,说明MALDI-TOF MS技术能区分同种菌株间的微小差异,根据菌株来源,推测优势群Ⅴ中14株野生菌株传播途径为草→进风口→取风室→采样人员→实验室→排风口,表明MALDI-TOF MS技术对阪崎克罗诺杆菌分型溯源具有重要价值。吕佳等[14]利用MALDI-TOF MS技术对4株肠炎沙门菌进行聚类分析,并将其结果与主成分分析结果进行比较,得出MALDI-TOF MS技术可为沙门菌分型溯源提供一种新的手段,由于本研究中试验菌株的数量有限,因此在这方面还需做进一步的探究。
VITEK MS与MALDI 7090的质谱图比对的结果表明,两者特征峰出峰位置与相对峰强度无太大差异,证明了基质辅助激光解吸的可靠性,由于MALDI 7090没有微生物数据库,并且获得的图谱无法转换为txt文件,因此只能比较两者谱图的异同,两者对微生物鉴定结果的异同还有待考察。
综上,MALDI-TOF MS技术应用于微生物的鉴定及溯源具有快速、简便和准确的优点,但也存在一些影响鉴定结果准确性和重复性的因素,另外,数据库中菌株信息的不全面和具有高效分辨力软件的缺乏,也是目前MALDI-TOF MS技术面临的挑战[15];因此,最大限度的发挥MALDI-TOF MS技术的优势,确保鉴定结果的准确性和可靠性,对于食源性致病菌的检测与鉴定、分型溯源等都具有重大意义。
参考文献
[1]BLACKWOOD B P, HUNTER C J. Cronobacter spp [J]. Microbiol Spectr, 2016, 4(2): 255-263.
[2]URMENYI A M C, FRANKLIN A W. Neonatal death from pigmented coliform infection [J]. Lancet, 1961, 1(7172):313-315.
[3]HOLY O, FORSYTHE S. Cronobacter spp. as emerging causes of healthcare-associated infection [J]. Journal of Hospital Infection, 2014, 86(3):169-177.
[4]HUNTER C J, BEAN J F. Cronobacter: an emerging opportunistic pathogen associated with neonatal meningitis, sepsis and necrotizing enterocolitis[J]. Journal of Perinatology Official Journal of the California Perinatal Association, 2013, 33(8):581-585.
[5]IVERSEN C, MULLANE N, MCCARDELL B, et al. Cronobacter gen. nov., a new genus to accommodate the biogroups of Enterobacter sakazakii, and proposal of Cronobacter sakazakii gen. nov., comb. nov., Cronobacter malonaticus sp. nov., Cronobacter turicensis sp. nov., Cronobacter muytjensii sp. nov., Cronobacter dublinensis sp.nov., Cronobacter genomospecies l, and of three subspecies, Cronobacter dublinensis subsp.dublinensis subsp. nov., Cronobacter dublinensis subsp. lausannensis subsp. nov. and Cronobacter dublinensis subsp. lactaridi subsp. nov. [J]. International Journal of Systematic & Evolutionary Microbiology, 2008, 58(6):1442-1447.
[6]GIOVANNINI M, VERDUCI E, GHISLENI D, et al. Enterobacter sakazakii: an emerging problem in paediatric nutrition [J]. Journal of International Medical Research, 2008, 36(3):394-399.
[7]HUNTER C J, PETROSYAN M, FORD H R, et al. Enterobacter sakazakii: an emerging pathogen in infants and neonates[J]. Surgical Infections, 2008, 9(5):533-539.
[8]其布勒哈斯. 应用MALDI-TOF质谱技术分析和鉴定食源性致病菌研究[D]. 呼和浩特:内蒙古农业大学, 2009.
[9]KASSIM A, PFLGER V, PREMJI Z, et al. Comparison of biomarker based matrix assisted laser desorption ionization-time of flight mass spectrometry (MALDI-TOF MS) and conventional methods in the identification of clinically relevant bacteria and yeast[J]. Bmc Microbiology, 2017, 17(1):128-136.
[10]CHO Y, KIM E, HAN S K, et al. Rapid identification of Vibrio species isolated from the southern coastal regions of Korea by MALDI-TOF mass spectrometry and comparison of MALDI sample preparation methods [J]. Journal of Microbiology & Biotechnology, 2017, 27(9):1593-1601.
[11]赵贵明, 刘洋, 陈颖,等. 克罗诺杆菌MALDI-TOF-MS数据库的建立及应用[J]. 食品科学, 2014, 35(8):105-110.
[12]李修远, 黄艳飞, 尚军, 等. MALDI-TOF MS在临床微生物鉴定中的常见问题及对策[J]. 临床检验杂志, 2016, 34(12):885-891.
[13]QI W, ZHAO X J, WANG Z W, et al. Identification of Cronobacter, species by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry with an optimized analysis method[J]. Journal of Microbiological Methods, 2017, 139(5):172-180.
[14]吕佳, 卢行安, 刘淑艳,等. MALDI-TOF-MS鉴定2008年辽宁省食源性疾病监测系统检出的沙门菌的初步研究[J]. 中国食品卫生杂志, 2011, 23(1):61-69.
[15]RAHI P, PRAKASH O, SHOUCHE Y S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists[J]. Frontiers in Microbiology, 2016, 7(4):1359-1371.
[2]URMENYI A M C, FRANKLIN A W. Neonatal death from pigmented coliform infection [J]. Lancet, 1961, 1(7172):313-315.
[3]HOLY O, FORSYTHE S. Cronobacter spp. as emerging causes of healthcare-associated infection [J]. Journal of Hospital Infection, 2014, 86(3):169-177.
[4]HUNTER C J, BEAN J F. Cronobacter: an emerging opportunistic pathogen associated with neonatal meningitis, sepsis and necrotizing enterocolitis[J]. Journal of Perinatology Official Journal of the California Perinatal Association, 2013, 33(8):581-585.
[5]IVERSEN C, MULLANE N, MCCARDELL B, et al. Cronobacter gen. nov., a new genus to accommodate the biogroups of Enterobacter sakazakii, and proposal of Cronobacter sakazakii gen. nov., comb. nov., Cronobacter malonaticus sp. nov., Cronobacter turicensis sp. nov., Cronobacter muytjensii sp. nov., Cronobacter dublinensis sp.nov., Cronobacter genomospecies l, and of three subspecies, Cronobacter dublinensis subsp.dublinensis subsp. nov., Cronobacter dublinensis subsp. lausannensis subsp. nov. and Cronobacter dublinensis subsp. lactaridi subsp. nov. [J]. International Journal of Systematic & Evolutionary Microbiology, 2008, 58(6):1442-1447.
[6]GIOVANNINI M, VERDUCI E, GHISLENI D, et al. Enterobacter sakazakii: an emerging problem in paediatric nutrition [J]. Journal of International Medical Research, 2008, 36(3):394-399.
[7]HUNTER C J, PETROSYAN M, FORD H R, et al. Enterobacter sakazakii: an emerging pathogen in infants and neonates[J]. Surgical Infections, 2008, 9(5):533-539.
[8]其布勒哈斯. 应用MALDI-TOF质谱技术分析和鉴定食源性致病菌研究[D]. 呼和浩特:内蒙古农业大学, 2009.
[9]KASSIM A, PFLGER V, PREMJI Z, et al. Comparison of biomarker based matrix assisted laser desorption ionization-time of flight mass spectrometry (MALDI-TOF MS) and conventional methods in the identification of clinically relevant bacteria and yeast[J]. Bmc Microbiology, 2017, 17(1):128-136.
[10]CHO Y, KIM E, HAN S K, et al. Rapid identification of Vibrio species isolated from the southern coastal regions of Korea by MALDI-TOF mass spectrometry and comparison of MALDI sample preparation methods [J]. Journal of Microbiology & Biotechnology, 2017, 27(9):1593-1601.
[11]赵贵明, 刘洋, 陈颖,等. 克罗诺杆菌MALDI-TOF-MS数据库的建立及应用[J]. 食品科学, 2014, 35(8):105-110.
[12]李修远, 黄艳飞, 尚军, 等. MALDI-TOF MS在临床微生物鉴定中的常见问题及对策[J]. 临床检验杂志, 2016, 34(12):885-891.
[13]QI W, ZHAO X J, WANG Z W, et al. Identification of Cronobacter, species by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry with an optimized analysis method[J]. Journal of Microbiological Methods, 2017, 139(5):172-180.
[14]吕佳, 卢行安, 刘淑艳,等. MALDI-TOF-MS鉴定2008年辽宁省食源性疾病监测系统检出的沙门菌的初步研究[J]. 中国食品卫生杂志, 2011, 23(1):61-69.
[15]RAHI P, PRAKASH O, SHOUCHE Y S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists[J]. Frontiers in Microbiology, 2016, 7(4):1359-1371.
 王伟,赵红阳,卢雁,王鸣雨,孙英健,宋娇娇,张顺合,卢行安.基质辅助激光解吸电离飞行时间质谱技术对阪崎克罗诺杆菌的鉴定及溯源[J].中国食品卫生杂志,2018,30(2):125-131.
王伟,赵红阳,卢雁,王鸣雨,孙英健,宋娇娇,张顺合,卢行安.基质辅助激光解吸电离飞行时间质谱技术对阪崎克罗诺杆菌的鉴定及溯源[J].中国食品卫生杂志,2018,30(2):125-131.