DOi:10.13590/j.cjfh.2017.05.001
产CTX-M-55型超广谱β-内酰胺酶大肠埃希菌全基因组序列耐药和毒力特征研究
(国家食品安全风险评估中心 卫生部食品安全风险评估重点实验室,北京 100021)
王伟 男 助理研究员 研究方向为食品微生物 E-mail:wangweiwsw@cfsa.net.cn 李凤琴 女 研究员 研究方向为食品微生物 E-mail:lifengqin@cfsa.net.cn
2017-09-13
国家重点研发计划“跨境食品潜在、新发病原微生物筛查监控技术及溯源平台建设”(2016YFD0401102);国家食品安全风险评估中心青年科研基金项目“肠道菌群介导小鼠抵抗鸡蛋致敏模型的建立及调控机制研究”(2017004)
摘要: 目的 基于全基因组序列,对2株食源性产CTX-M-55型超广谱β-内酰胺酶(ESBLs)大肠埃希菌的耐药和毒力分子机制进行研究。方法 采用微量肉汤稀释法对大肠埃希菌食品分离株进行药敏试验,设计产ESBLs基因引物并通过聚合酶链式反应(PCR)扩增筛选出2株产CTX-M-55型ESBLs大肠埃希菌(编号为EC001和EC002)进行全基因组测序,并进行序列型(ST)、质粒复制子类型、血清型、耐药基因和毒力因子识别。结果 2株大肠埃希菌均为头孢类和喹诺酮类抗生素双重耐药ESBLs菌株;全基因组序列分析结果显示,2株菌的血清型均为肠道致病性大肠埃希菌(EPEC)O119∶H8,ST型分别为ST21(EC001)和ST342(EC002),EC001菌株含有IncFII、IncX1、IncY、Col156和IncI2 5个不相容群质粒,EC002菌株含有IncFII和IncX1 2个不相容群质粒,2株菌株染色体中均携带blaCTX-M-55和blaTEM-1412种ESBLs基因。此外,EC001菌株还携带sul2/3、tet(A)/(B)、dfrA12、strA/B、aph(3′)-IIa、cml/cmlA1、floR和oqxA/B耐药相关基因,其染色体喹诺酮耐药决定区gyrA基因存在2个突变位点(S83L,D87H)、parC基因存在1个突变位点(S80I),多粘菌素耐药相关基因pmrE基因存在3个突变位点(D73Y,M185T,S225T);而EC002菌株携带fosA和qnrS1耐药相关基因。2株菌均携带致黏附与脱落(A/E)损伤的肠细胞脱落位点(LEE)毒力岛基因esc、esp、eaeA和tir,且EC001菌株还携带增强菌株在血清中存活能力的iss基因。结论 对2株食源性产CTX-M-55型ESBLs大肠埃希菌进行全基因组测序以及耐药和毒力的分子机制的研究,其结果可为后续开展大肠埃希菌耐药和毒力表型预测、指导食用畜禽养殖过程中抗生素合理使用及耐药菌株防控提供依据。
关键词:
全基因组测序; CTX-M-55型; 超广谱β-内酰胺酶; 耐药; 大肠埃希菌
中图分类号: R155 文献标识码:A 文章编号:1004-8456(2017)05-0519-07
Genetic characterization of two CTX-M-55 type extended-spectrum
β-lactamase-producing Escherichia coli isolates
β-lactamase-producing Escherichia coli isolates
(Key Laboratory of Food Safety Risk Assessment of Ministry of Health,China National Center for Food Safety Risk Assessment,Beijing 100021,China)
Abstract: Objective To study the antimicrobial resistance and virulence mechanism of two foodborne CTX-M-55 type extended-spectrum β-lactamase (ESBL) -producing Escherichia coli strains based on whole genome sequences. Methods
The antimicrobial susceptibility testing of two CTX-M-55 type ESBL-producing Escherichia coli isolates (EC001 and EC002) that were collected from foodborne pathogen surveillance system in 2011 was conducted by antimicrobial susceptibility test and PCR. Then two strains were sequenced using the Illunima Hiseq 2000 platform. Based on the assembled genome sequences of two strains, the annotation of sequence type, plasmid replicon type, serotype, and antimicrobial resistance/virulence genes was performed using BioNumerics software. Results The two strains were identified to be ESBL-producing and fluoroquinolone-resistant. The genomic analysis result showed that strains EC001 and EC002 were ST21 and ST342, respectively, and serotyped to be enteropathogenic Escherichia coli (EPEC) O119∶ H8. The plasmid of EC001 belonged to 5 incompatible groups including IncFII, IncX1, IncY, Col156, and IncI2, and that of EC002 belonged to 2 incompatible groups including IncFII and IncX1. Notably, both strains harbored 2 types of ESBL-encoding genes blaCTX-M-55 and blaTEM-141. Additionally, the multi-antimicrobial resistance genes sul2/3, tet(A) / (B), dfrA12, strA/B, aph(3′)-IIa, cml/cmlA1, floR, and oqxA/B were identified on the genome of strain EC001, while fosA and qnrS1 were identified on the genome of strain EC002. Compared with the genome of Escherichia coli K12_ 1655, two mutations were found at the 83th (S83L) and 87th (D87H) codons of gyrA, and one mutation was found at the 80th (S80I) codon of parC in QRDRs of strain EC001. Noteworthy, 3 mutations were found at the 73th (D73Y), 185th (M185T) and 225th (S225T) codons of pmrE in the chromosome of strain EC001, which might be responsible for its resistance to colistin. The virulence gene prediction result showed that both strains harbored classical locus of enterocyte and effacement (LEE) genes esc, esp, eaeA and tir, while EC001 contained iss gene which could increase serum survival capacity of this bacterium. Conclusion To our best knowledge, we applied whole genome sequencing in annotation of the resistance and virulence mechanism of two CTX-M-55-type ESBL-producing Escherichia coli strains in China. The data in this study will shed light on antimicrobial resistance and virulence mechanisms of Escherichia coli,and finally help prevent and control the contamination of this foodborne pathogen.
The antimicrobial susceptibility testing of two CTX-M-55 type ESBL-producing Escherichia coli isolates (EC001 and EC002) that were collected from foodborne pathogen surveillance system in 2011 was conducted by antimicrobial susceptibility test and PCR. Then two strains were sequenced using the Illunima Hiseq 2000 platform. Based on the assembled genome sequences of two strains, the annotation of sequence type, plasmid replicon type, serotype, and antimicrobial resistance/virulence genes was performed using BioNumerics software. Results The two strains were identified to be ESBL-producing and fluoroquinolone-resistant. The genomic analysis result showed that strains EC001 and EC002 were ST21 and ST342, respectively, and serotyped to be enteropathogenic Escherichia coli (EPEC) O119∶ H8. The plasmid of EC001 belonged to 5 incompatible groups including IncFII, IncX1, IncY, Col156, and IncI2, and that of EC002 belonged to 2 incompatible groups including IncFII and IncX1. Notably, both strains harbored 2 types of ESBL-encoding genes blaCTX-M-55 and blaTEM-141. Additionally, the multi-antimicrobial resistance genes sul2/3, tet(A) / (B), dfrA12, strA/B, aph(3′)-IIa, cml/cmlA1, floR, and oqxA/B were identified on the genome of strain EC001, while fosA and qnrS1 were identified on the genome of strain EC002. Compared with the genome of Escherichia coli K12_ 1655, two mutations were found at the 83th (S83L) and 87th (D87H) codons of gyrA, and one mutation was found at the 80th (S80I) codon of parC in QRDRs of strain EC001. Noteworthy, 3 mutations were found at the 73th (D73Y), 185th (M185T) and 225th (S225T) codons of pmrE in the chromosome of strain EC001, which might be responsible for its resistance to colistin. The virulence gene prediction result showed that both strains harbored classical locus of enterocyte and effacement (LEE) genes esc, esp, eaeA and tir, while EC001 contained iss gene which could increase serum survival capacity of this bacterium. Conclusion To our best knowledge, we applied whole genome sequencing in annotation of the resistance and virulence mechanism of two CTX-M-55-type ESBL-producing Escherichia coli strains in China. The data in this study will shed light on antimicrobial resistance and virulence mechanisms of Escherichia coli,and finally help prevent and control the contamination of this foodborne pathogen.
Key words:
Whole genome sequencing; CTX-M-55; extended-spectrum β-lactamase; resistance; Escherichia coli
β-内酰胺酶是细菌产生的可以水解青霉素类、头孢类等抗生素的蛋白。该酶数量众多,在抗生素应用的压力下不断突变,导致超广谱β-内酰胺酶(extended-spectrum β-lactamase, ESBL)的形成。β-内酰胺酶分为染色体介导酶和耐药质粒介导酶两大类,质粒介导的β-内酰胺酶有TEM型、OXA型、CTX-M型、SHV-1型和HMS-1型等,携带ESBLs耐药基因的质粒在不同细菌种属间的水平转移被认为是多重耐药基因传播的重要机制之一,已成为全球重要的公共卫生问题[1]。 分子流行病学调查[2]显示,1980—2000年间我国多重耐药大肠埃希菌编码blaESBLs可转移质粒产ESBLs的基因型主要是TEM型和SHV型,而2000年以后,CTX-M已取代TEM和SHV成为我国最主要的ESBLs基因型,这一变化的产生一方面与blaCTX-M基因通过可移动基因元件如质粒和转座子等介导的菌株间高频率传递有关[3];另一方面,CTX-M型产ESBLs菌株可同时对喹诺酮类和氨基糖苷类抗生素表现为多重耐药,这种协同耐药性增强了该型大肠埃希菌对抗生素的抵抗性,从而导致其在环境或宿主体内的长期存在[4-5]。其中,blaCTX-M-55已成为我国人、动物和环境来源大肠埃希菌分离株中流行的主要CTX-M型别[4],给社区人群健康和临床治疗带来严重隐患[6-7]。我国虽有从食品及养殖动物中分离到产CTX-M-55型ESBLs大肠埃希菌的报道[8-10],但鲜有从全基因组水平对其耐药性和致病性进行分析。本研究通过二代测序技术对从食品中分离的2株产CTX-M-55型ESBLs大肠埃希菌进行全基因组测序,并在对测序数据进行拼接组装的基础上,对其耐药和致病机制进行全景式表征,旨在为食用畜禽养殖过程中抗生素的合理使用及耐药菌株污染防控提供参考。
Table 3 Genomic characterization and annotation of 2 E.coli strains
注:—表示未识别到该基因
注:A和B分别为菌株EC001和EC002的基因组圈图,每个圈图从内到外依次为GC含量、GC偏倚、
正义链、反义链和注释的功能基因,绿色字体为质粒复制子,红色字体为耐药基因,黑色字体为毒力基因
为2株菌共同携带的基因,其余基因均为EC001独有基因
通过与BioNumerics软件大肠埃希菌基因分型模块内置的耐药基因数据库比对,2株菌共识别到介导10类抗生素耐药的20个耐药基因(具体见表2、图1和图2)。其中EC001菌株基因组中共识别到介导9类抗生素耐药的18个耐药基因,包括介导磺胺类抗生素(磺胺甲恶唑)耐药的sul2/3、介导四环素类抗生素耐药的tet(A)/(B)、介导甲氧苄啶类抗生素(甲氧苄啶)耐药的dfrA12、介导氨基糖苷类抗生素耐药的strA/B和aph(3′)-IIa、介导β-内酰胺酶类抗生素耐药的blaCTX-M-55和blaTEM-141、介导氯霉素类抗生素耐药的cml/cmlA1和floR等。EC002菌株基因组中共识别到介导5类抗生素耐药的4个耐药基因,包括介导青霉素类、头孢类、β-内酰胺酶类抗生素耐药的blaCTX-M-55、blaTEM-141和介导喹诺酮类抗生素耐药的qnrS1以及介导磷霉素耐药的fosA。与大肠埃希菌K12_1655菌株QRDRs拓扑异构酶编码基因序列的比对发现,EC001菌株的染色体QRDRs存在突变,分别为gyrA第83位由丝氨酸突变为亮氨酸(S83L)、第87位由天冬氨酸突变为组氨酸(D87H)以及parC第80位丝氨酸突变为异亮氨酸(S80I),详见表2。同时,与大肠埃希菌K12_1655菌株染色体上编码多粘菌素耐药相关基因的比对发现,EC001菌株的pmrE基因存在3个突变位点,分别为第73位由天冬氨酸突变为酪氨酸(D73Y)、第185位由甲硫氨酸突变为苏氨酸(M185T)和第225位由丝氨酸突变为苏氨酸(S225T),详见表2。与软件大肠埃希菌基因分型模块内置毒力基因数据库比对结果表明,2株菌均携带致黏附与脱落(adherence/effacement,A/E)损伤的肠细胞脱落位点(locus of enterocyte and effacement, LEE,又称LEE毒力岛),主要包括编码Ⅲ型分泌系统esc基因、分泌型蛋白质esp基因、外膜蛋白紧密素eaeA基因和转位紧密粘附素受体tir基因等,此外在EC001染色体中还识别到增强菌株在血清中存活能力的iss基因,具体详见表3和图1。
1 材料与方法
1.1 材料
1.1.1 菌株来源
以我国食品安全风险监测网从食品中分离到的50株食源性大肠埃希菌为研究对象,菌株初步鉴定为致泻大肠埃希菌[11],于40%甘油的脑心浸液肉汤冻存管中,-80 ℃冰箱储存备用。
1.1.2 主要仪器与试剂
Illumina Hiseq 2000测序系统(美国Illumina),NanoDrop Lite紫外分光光度计(美国Thermo-Fisher),全基因组DNA提取试剂盒(德国Qiagen)。T载体、感受态细胞均购自日本TAKAR,试验用抗生素均购自美国Sigma。
1.2 方法
1.2.1 抗生素敏感性试验
根据美国临床和实验室标准委员会(Clinical and Laboratory Standards Institute,CLSI)推荐的微量肉汤稀释法[12],以大肠埃希菌(ATCC 25922)作为质控菌株,测定17种抗生素对受试菌株的最低抑菌浓度(minimum inhibitory concentration,MIC),参考CLSI和美国国家抗菌素耐药监测系统(National Antimicrobial Resistance Monitoring System,NARMS)相关标准对耐药折点进行判读。若菌株对头孢他啶耐药,则按照CLSI推荐的琼脂稀释法对其进行ESBLs表型确证。确证试验中若头孢他啶/克拉维酸配伍对受试菌株的MIC值较头孢他啶单独试验的MIC值降低了8倍及以上时,判定为ESBLs阳性。
1.2.2 ESBLs分型
对1.2.1中筛选出的菌株进行ESBLs blaCTX-M分析[13],先确定CTX组别,再用blaCTX-M 亚分组(subgroup)进行分型[14-15]。对阳性扩增产物,将纯化后的聚合酶链式反应(PCR)产物连接至T载体,并转至感受态细胞,操作步骤参照试剂盒说明书,送赛默飞世尔公司测序,将测序结果在美国国家生物技术信息中心(NCBI)BLAST上进行比对获得亚分型,同时对菌株进行ESBLs blaTEM分析[16]。引物序列见表1。试验共筛选出2株产CTX-M-55型ESBLs大肠埃希菌,分别编号为EC001和EC002,于40%甘油的脑心浸液肉汤冻存管中,-80 ℃冰箱储存备用。
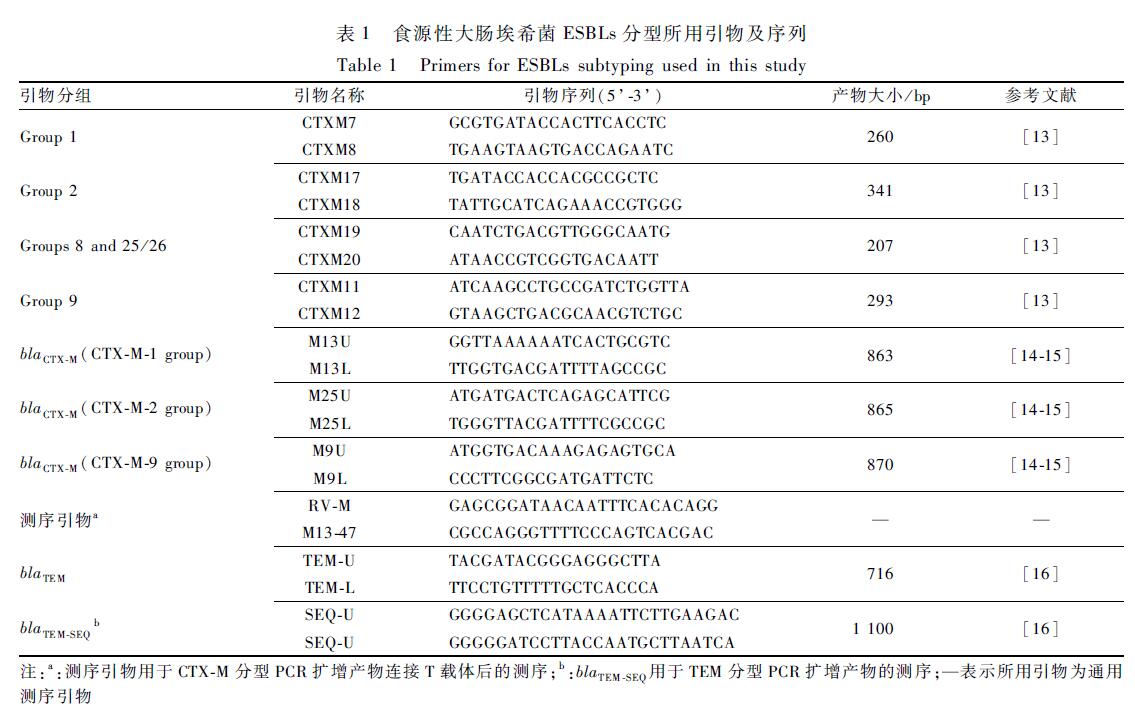
|
表1 食源性大肠埃希菌ESBLs分型所用引物及序列 Table 1 Primers for ESBLs subtyping used in this study 注:a:测序引物用于CTX-M分型PCR扩增产物连接T载体后的测序;b:blaTEM-SEQ用于TEM分型PCR扩增产物的测序;—表示所用引物为通用测序引物 |
1.2.3 全基因组DNA提取
从冻存管中分别挑取1.2.2筛选出的2株产ESBLs大肠埃希菌,划线接种于脑心浸液琼脂平板,(37±1) ℃培养18~24 h后,挑取单菌落转种于10 ml脑心浸液肉汤,(37±1) ℃培养18~24 h后,按试剂盒说明书操作提取基因组DNA,提取的基因组DNA浓度要求>50 ng/ml,总量均>2 μg,并经0.8%琼脂糖凝胶电泳检测验证。
1.2.4 全基因组序列测定
2株大肠埃希菌基因组提取产物送往北京诺禾致源科技股份有限公司进行全基因组序列测定。采用超声法将2株大肠埃希菌基因组大片段DNA随机打断并产生主带≤800 bp的一系列DNA片段,用T4 DNA聚合酶、DNA聚合酶和T4多聚核酸激酶将打断形成的DNA粘性末端修复成平末端,再通过3′端加碱基腺嘌呤(A),使得DNA片段能与3′端带有胸腺嘧啶(T)碱基的特殊接头连接,用电泳法选择需回收的目的片段连接产物,再使用PCR技术扩增两端带有接头的DNA片段;最后进行文库构建和Illumina Hiseq 2000平台测序。
1.2.5 基因组组装及注释
通过BioNumerics V7.6软件内置全基因组测序数据生物信息学分析流程进行2株产CTX-M-55型大肠埃希菌基因组组装及注释。每株菌原始测序数据读长各碱基质量值(Q值)的平均值均>30,序列拼接组装基于全基因组多位点序列分型(wgMLST)内置从头组装(de novo assemble)算法完成,序列型(sequence type,ST)等位基因号基于wgMLST内置自动组装(assembly free calls)算法获取;质粒复制子类型、血清型、耐药基因和毒力基因的识别基于软件大肠埃希菌基因分型(E.coli genotyping)模块内置数据库完成。此外,通过待测菌株序列与大肠埃希菌K12_1655(GenBank:NC_000913)染色体喹诺酮耐药决定区(QRDRs)拓扑异构酶II的gyrA和gyrB序列、拓扑异构酶IV的parC和parE序列以及染色体编码多粘菌素耐药的phoPQ系统、pmrAB双分系统、pmrHFUKLM操纵子以及pmrE基因序列的比对,确定本研究2株菌的喹诺酮及多粘菌素耐药突变位点。
2结果
2.1药敏结果
ESBLs分型试验共从50株食源性大肠埃希菌获得2株产CTX-M-55型ESBLs大肠埃希菌,其药敏结果见表2。由表可见,2株菌均为头孢类和喹诺酮类抗生素双重耐药菌株,其中EC001菌株除对美罗培南(碳青霉烯类)、庆大霉素(氨基糖苷类)和阿奇霉素(大环内酯类)抗生素敏感外,对其他所有测试的抗生素均耐药;EC002菌株则对氨苄西林(青霉素类)、头孢他啶(头孢类)、阿莫西林/克拉维酸(β-内酰胺酶类抑制剂)、萘啶酸和环丙沙星(喹诺酮类)耐药。2株菌均为产ESBLs菌株。
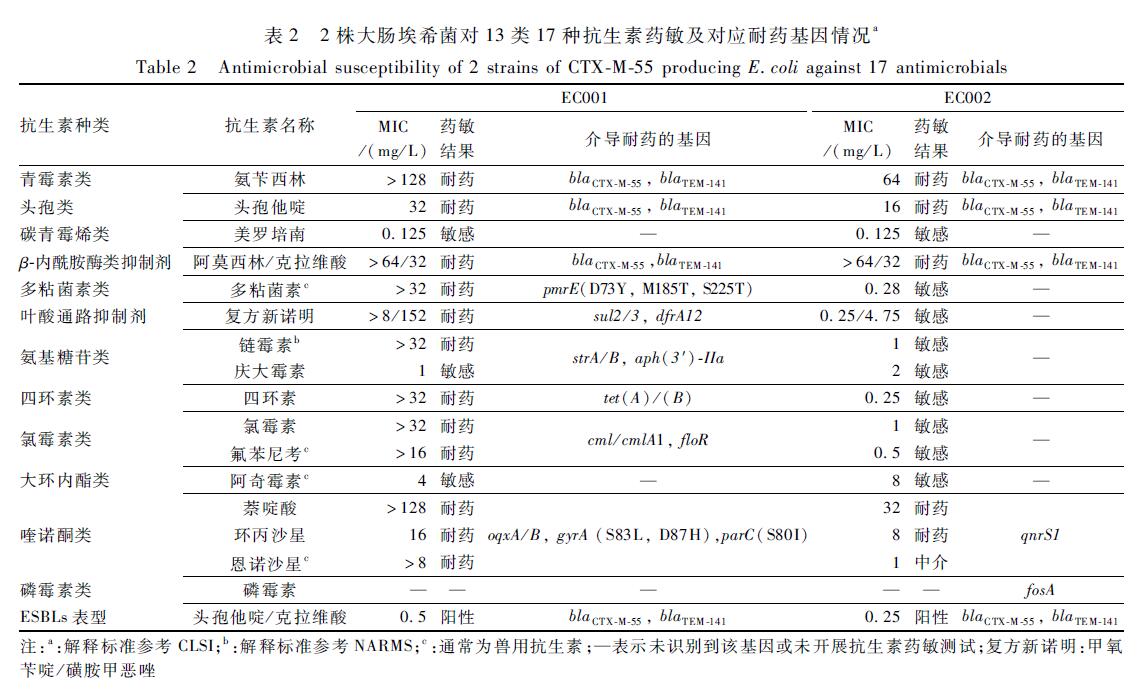
|
表2 2株大肠埃希菌对13类17种抗生素药敏及对应耐药基因情况a Table 2 Antimicrobial susceptibility of 2 strains of CTX-M-55 producing E.coli against 17 antimicrobials 注:a:解释标准参考CLSI;b:解释标准参考NARMS;c:通常为兽用抗生素;—表示未识别到该基因或未开展抗生素药敏测试;复方新诺明:甲氧苄啶/磺胺甲恶唑 |
2.2 基因组序列组装结果
用BioNumerics软件对2株菌基因组序列数据进行拼接组装,结果显示,EC001菌株基因组大小为4.80 Mb,鸟嘌呤和胞嘧啶所占的比率(GC含量)为50.72%,含有4 691个基因,总长度4 799 116 bp,平均长度1 023 bp,占基因组全长的89.36%,含有RNA 74个;EC002菌株基因组大小为4.45 Mb,GC含量为50.89%,含有4 272个基因,总长度4 447 148 bp,平均长度1 041 bp,占基因组全长的88.95%,含有RNA 83个。详见表3和图1。表3 2株大肠埃希菌基因组注释结果Table 3 Genomic characterization and annotation of 2 E.coli strains
注:—表示未识别到该基因
注:A和B分别为菌株EC001和EC002的基因组圈图,每个圈图从内到外依次为GC含量、GC偏倚、
正义链、反义链和注释的功能基因,绿色字体为质粒复制子,红色字体为耐药基因,黑色字体为毒力基因
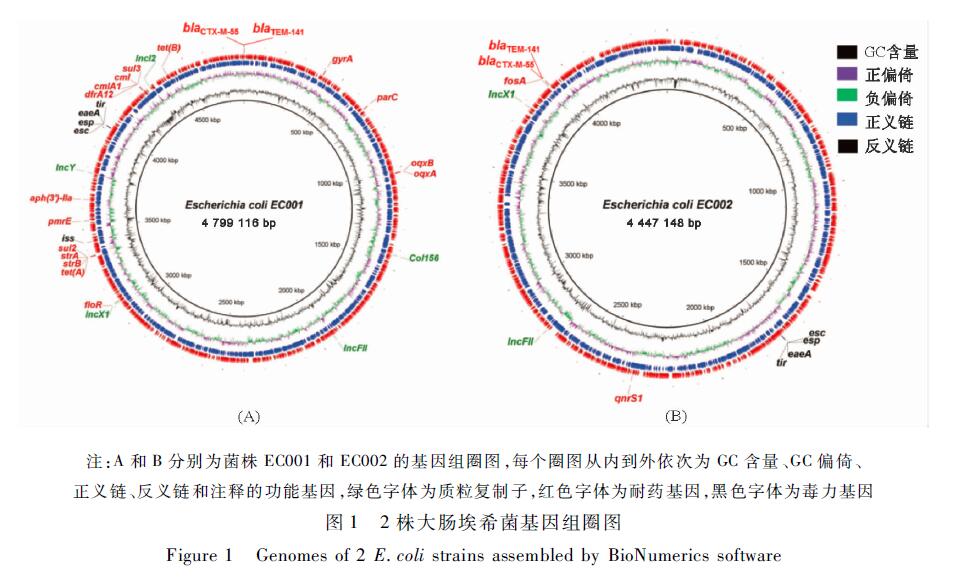
|
图1 2株大肠埃希菌基因组圈图 Figure 1 Genomes of 2 E.coli strains assembled by BioNumerics software |
2.3 全基因组序列的注释结果
经BioNumerics软件的注释结果表明,2株菌的血清型均为O119∶H8,属于肠道致病性大肠埃希菌(Enteropathogenic Escherichia coli,EPEC),EC001和EC002菌株的ST型分别为ST21和ST342。其中EC001菌株携带的质粒属于5个不相容群,即IncFII、IncX1、IncY、Col156和IncI2,EC002菌株携带的质粒属于2个不相容群,即IncFII和IncX1,详见表3和图1。
2.4 耐药和毒力基因注释结果
注:qnrS1和fosA为EC002独有基因,blaCTX-M-55和blaTEM-141为2株菌共同携带的基因,其余基因均为EC001独有基因

|
图2 2株大肠埃希菌抗生素耐药基因热图 Figure 2 Heat map of antibiotic resistance ontology categories and antibiotic resistance genes on 2 E.coli strains |
3 讨论
自1990年产CTX-M型ESBLs大肠埃希菌首次报道以来[17],世界各国食用畜禽及人类感染产CTX-M型ESBLs大肠埃希菌的情况日益严重,我国社区人群和食用畜禽中产CTX-M-55型ESBLs大肠埃希菌的检出率自2011年以来也呈逐年上升趋势[18]。本研究结果显示,除EC002菌株对恩诺沙星表现为中介外,2株菌几乎对测试的所有头孢类和喹诺酮类抗生素均耐药。环丙沙星和头孢类抗生素是临床治疗肠杆菌科感染的首选用药,而恩诺沙星又是养殖动物专用的喹诺酮类抗生素[19-21],人或动物一旦感染上述产CTX-M-55型ESBLs大肠埃希菌,极有可能导致治疗失败。此外,EC001菌株对多粘菌素、链霉素、四环素、氯霉素和氟苯尼考等均表现为较高的耐药水平,而多粘菌素和氟苯尼考是我国肉猪养殖中常用的饲料添加剂[22-23],提示上述产CTX-M-55型ESBLs大肠埃希菌已成为威胁我国居民健康和生命安全的重要食源性致病菌,因此在基因水平对其耐药机制的基础研究,是发展耐药菌有效防控措施的关键。 研究[24-25]显示,blaCTX-M-55可位于相同或异种复制子类型的质粒上进行传播。本研究2株产CTX-M-55型ESBLs大肠埃希菌携带有5种不同的复制子类型(IncFII、IncX1、IncY、Col156、IncI2),其中IncFII和IncI2是近年来报道较多的复制子类型[26],提示该2株菌携带的blaCTX-M-55耐药基因可随IncFII和IncI2型质粒在动物源大肠埃希菌及其他菌属间传播,并通过食物链传递给人类。 本研究采用BioNumerics软件内置的耐药基因数据库,对2株大肠埃希菌基因组中耐药基因进行了识别。结果显示2株产CTX-M-55型ESBLs大肠埃希菌均识别到blaCTX-M-55和blaTEM-141基因,这与菌株对头孢类和青霉素类抗生素的耐药表型及PCR扩增获得的基因型完全符合。喹诺酮类抗生素耐药机制中gyrA和parC的点突变是最常见的[27],本研究通过对2株菌染色体比对发现,EC001菌株gyrA存在两个点突变S83L和D87H,parC存在一个点突变S80I。此外,在2株菌中还识别到质粒介导的喹诺酮耐药基因(plasmid-mediated quinolone resistance,PMQR)[28],分别为oqxA/B和qnrS1基因。EC001还携带磺胺类抗生素耐药基因sul2/3和甲氧苄啶耐药基因dfrA12,菌株获得sul2基因后可编码产生磺胺类抗生素所无法抑制的二氢叶酸合成酶来介导耐药[29],而sul3则是一种新型的质粒介导的磺胺类抗生素耐药基因,多出现在携带I类整合子的耐药质粒上,通常也会带有aadA和dfrA(包括dfrA12基因)基因盒,可调控菌株对复方新诺明的耐药[30]。EC001还携带四环素类耐药基因tet(A)/(B),在介导四环素类耐药的7个tet基因中,tet(A)基因是大肠埃希菌菌株中出现频率最高的,其次为tet(B)基因,并且tet(A)在动物来源耐药大肠埃希菌中的检出率呈逐年上升趋势[31]。EC001还携带氨基糖苷类抗生素耐药基因strA/B和aph(3′)-IIa,均为耐药大肠埃希菌最常见的修饰酶,其中strA/B基因介导链霉素耐药,而aph(3′)-IIa基因则主要介导卡那霉素和新霉素耐药[32-33],未识别到介导庆大霉素耐药的aac(3)-IIa基因[34],这与EC001对所测试的链霉素耐药而庆大霉素敏感的表型试验相符。EC001还携带氯霉素类抗生素耐药基因cml/cmlA1和floR,cml和cmlA1编码的细胞内膜cml蛋白,通过介导主动外排系统可使氯霉素在菌体内的含量明显减少从而实现耐药[35],而floR基因介导的大肠埃希菌对氟苯尼考的耐药性,在经济型动物中日益严重,已成为国内外的研究热点[36]。值得注意的是,在EC001染色体中还识别到与多粘菌素耐药相关的pmrE基因存在3个突变位点(D73Y,M185T,S225T),pmrE基因上有脂多糖(lipolpolysaccharide, LPS)修饰酶,可使菌株对多粘菌素类药物敏感度降低[37]。此外,EC001菌株中还识别与保护菌株抵抗血清杀灭作用相关的iss基因,说明该菌株感染人类后在血液中可以抵抗来自机体的杀伤作用[36]。研究数据可为开展大肠埃希菌血清型鉴定、耐药表型和毒力表型等的预测、耐药和毒力机制解析提供新思路,同时可为指导食用畜禽动物养殖过程中抗生素的合理使用及耐药菌株污染防控提供依据。
参考文献
[1]BUYNAK J D.Understanding the longevity of the beta-lactam antibiotics and of antibiotic/beta-lactamase inhibitor combinations[J].Biochem Pharmacol,2006,71(7):930-940.
[2]YU Y S,JI S J,CHEN Y G,et al.Resistance of strains producing extended spectrum β-lactamases and genotype distribution in China[J].J Infect,2007,54(1):53-57.
[3]CANTN R,GONZLEZ-ALBA J M,GALN J C.CTX-M enzymes:origin and diffusion[J].Front Microbiol,2012,3:110.
[4]D’ANDREA M M,ARENA F,PALLECCHI L,et al.CTX-M-type beta-lactamases:a successful story of antibiotic resistance[J].Int J Med Microbiol,2013,303(6/7):305-317.
[5]CANTN R,COQUE T M,BAQUERO F.Multi-resistant Gram-negative bacilli:from epidemics to endemics[J].Curr Opin Infect Dis,2003,16(4):315-325.
[6]MEMARIANI M,NAJAR P S,ZAHRAEI S T,et al.Occurrence of SHV,TEM and CTX-M β-gactamase genes among enteropathogenic Escherichia coli strains isolated from children with diarrhea[J].Jundishapur J Microbiol,2015,8(4):e15620.
[7]SCALETSKY I C,SOUZA T B,ARANDA K R,et al.Genetic elements associated with antimicrobial resistance in enteropathogenic Escherichia coli(EPEC)from Brazil[J].BMC Microbiol,2010,10:25.
[8]LI J Y,MA Y,HU C Q,et al.Dissemination of cefotaxime-M-producing Escherichia coli isolates in poultry farms,but not swine farms,in China[J].Foodborne Pathog Dis,2010,7(11):1387-1392.
[9]RAO L L,LYU L C,ZENG Z L,et al.Increasing prevalence of extended-spectrum cephalosporin-resistant Escherichia coli in food animals and the diversity of CTX-M genotypes during 2003-2012[J].Vet Microbiol,2014,172(3/4):534-541.
[10]XU G F,AN W,WANG H D,et al.Prevalence and characteristics of extended-spectrum β-lactamase genes in Escherichia coli isolated from piglets with post-weaning diarrhea in Heilongjiang province,China[J].Front Microbiol,2015,6:1103.
[11]中华人民共和国国家卫生和计划生育委员会,国家食品药品监督管理总局.食品安全国家标准 食品微生物学检验 致泻大肠埃希氏菌检验:GB 4789.6—2016[S].北京:中国标准出版社,2016.
[12]Clinical Laboratory Standards Institute. Methods for antimicrobial dilution and disk susceptibility testing of infrequently isolated or fastidious bacteria; approved guideline[S]. Wayne Pennsylvania: Clinical Laboratory Standards Institute, 2015.
[13]XU L, ENSOR V M, GOSSAIN S, et al. Rapid and simple detection of blaCTX-M genes by multiplex PCR assay[J].J Med Microbiol, 2005, 54(pt 12):1183-1187.
[14]ECKERT C, GAUTIER V, SALADIN-ALLARD M, et al. Dissemination of CTX-M-type beta-lactamases among clinical isolates of Enterobacteriaceae in Paris, France[J]. Antimicrob Agents Chemother, 2004, 48(4):1249-1255.
[15]JIANG X F, ZHANG Z, LI M, et al. Detection of extended-spectrum β-lactamases in clinical isolates of Pseudomonas aeruginosa[J]. Antimicrob Agents Chemother 2006, 50(9):2990-2995.
[16]KARCZMARCZYK M, ABBOTT Y, WALSH C, et al. Characterization of multidrug-resistant Escherichia coli isolates from animals presenting at a university veterinary hospital[J]. Appl Environ Microbiol,2011, 77(20): 7104-7112.
[17]BAUERNFEIND A,JUNGWIRTH R,SCHWEIGHART S,et al.Antibacterial activity and beta-lactamase stability of eleven oral cephalosporins[J].Infection,1990,18(3):155-167.
[18]XIA S,FAN X,HUANG Z G,et al.Dominance of CTX-M-type extended-spectrum β-lactamase(ESBL)-producing Escherichia coli isolated from patients with community-onset and hospital-onset infection in China[J].PLoS One,2014,9(7):e100707.
[19]HOANG P H,AWASTHI S P,DO NGUYEN P,et al.Antimicrobial resistance profiles and molecular characterization of Escherichia coli strains isolated from healthy adults in Ho Chi Minh City,Vietnam[J].J Vet Med Sci,2017,79(3):479-485.
[20]黄耀凌,刘智宏,白玉惠,等.恩诺沙星注射液在猪体内的残留消除研究[J].中国兽药杂志,2017,51(3):48-54.
[21]丁丽玲,鲁晓旭,李银生,等.蚯蚓体内恩诺沙星残留的检测方法研究[J].农业环境科学学报,2016,3(2):403-408.
[22]王影,李艳然,韩镌竹,等.多粘菌素耐药性的研究进展[J].微生物学通报,2017,44(1):200-206.
[23]刘艳红,吕淑霞,李颖,等.大肠杆菌耐药性及氟苯尼考耐药基因floR的研究[J].科技学术,2017,53(1):115-118.
[24]XIA L,LIU Y, XIA S,et al.Prevalence of ST1193 clone and IncI1/ST16 plasmid in E-coli isolates carrying blaCTX-M-55 gene from urinary tract infections patients in China[J].Sci Rep,2017,7:44866.
[25]LYU L C,PARTRIDGE S R,HE L Y,et al.Genetic characterization of IncI2 plasmids carrying blaCTX-M-55 spreading in both pets and food animals in China[J].Antimicrob Agents Chemother,2013,57(6):2824-2827.
[26]CARATTOLI A.Plasmids in Gram negatives:molecular typing of resistance plasmids[J].Int J Med Microbiol,2011,301(8):654-658.
[27]MORGAN-LINNELL S K,BOYD L B,STEFFEN D,et al.Mechanisms accounting for fluoroquinolone resistance in Escherichia coli clinical isolates[J].Antimicrob Agents Chemother,2009,53(1):235-241.
[28]HANSEN L H,JOHANNESEN E,BURMLLE M,et al.Plasmid-encoded multidrug efflux pump conferring resistance to olaquindox in Escherichia coli[J].Antimicrob Agents Chemother,2004,48(9):3332-3337.
[29]ENNE V I,LIVERMORE D M,STEPHENS P,et al.Persistence of sulphonamide resistance in Escherichia coli in the UK despite national prescribing restriction[J].Lancet,2001,357(9265):1325-1328.
[30]AJIBOYE R M,SOLBERG O D,LEE B M,et al.Global spread of mobile antimicrobial drug resistance determinants in human and animal Escherichia coli and Salmonella strains causing community-acquired infections[J].Clin Infect Dis,2009,49(3):365-371.
[31]GUERRA B,JUNKER E,SCHROETER A,et al.Phenotypic and genotypic characterization of antimicrobial resistance in German Escherichia coli isolates from cattle,swine and poultry[J].J Antimicrob Chemother,2003,52(3):489-492.
[32]MOMTAZ H,RAHIMI E,MOSHKELANI S.Molecular detection of antimicrobial resistance genes in E.coli isolated from slaughtered commercial chickens in Iran[J].Vet Med(Praha),2012,57(4):193-197.
[33]NSOFOR C A,IROEGBU C U,CALL D R,et al.Detection of antibiotic resistance genes of Escherichia coli from domestic livestock in southeast Nigeria with DNA microarray[J].J Cell Anim Biol,2013,7(12):149-163.
[34]ZHANG F Y,HUO S Y,LI Y R,et al.A survey of the frequency of aminoglycoside antibiotic-resistant genotypes and phenotypes in Escherichia coli in broilers with septicaemia in Hebei,China[J].Br Poul Sci,2014,55(3):305-310.
[35]杜向党,阎若潜,沈建忠.氯霉素类药物耐药机制的研究进展[J].动物医学进展,2004,25(2):27-29.
[36]郑朝朝,刘聚祥,李辉,等.鸡源,猪源大肠杆菌氟苯尼考耐药基因floR的克隆与序列分析[J].华北农学报,2010,25(5):72-75.
[37]YAGUCHI K,OGITANI T,OSAWA R,et al.Virulence factors of avian pathogenic Escherichia coli strains isolated from chickens with colisepticemia in Japan[J].Avian Dis,2007,51(3):656-662.
[2]YU Y S,JI S J,CHEN Y G,et al.Resistance of strains producing extended spectrum β-lactamases and genotype distribution in China[J].J Infect,2007,54(1):53-57.
[3]CANTN R,GONZLEZ-ALBA J M,GALN J C.CTX-M enzymes:origin and diffusion[J].Front Microbiol,2012,3:110.
[4]D’ANDREA M M,ARENA F,PALLECCHI L,et al.CTX-M-type beta-lactamases:a successful story of antibiotic resistance[J].Int J Med Microbiol,2013,303(6/7):305-317.
[5]CANTN R,COQUE T M,BAQUERO F.Multi-resistant Gram-negative bacilli:from epidemics to endemics[J].Curr Opin Infect Dis,2003,16(4):315-325.
[6]MEMARIANI M,NAJAR P S,ZAHRAEI S T,et al.Occurrence of SHV,TEM and CTX-M β-gactamase genes among enteropathogenic Escherichia coli strains isolated from children with diarrhea[J].Jundishapur J Microbiol,2015,8(4):e15620.
[7]SCALETSKY I C,SOUZA T B,ARANDA K R,et al.Genetic elements associated with antimicrobial resistance in enteropathogenic Escherichia coli(EPEC)from Brazil[J].BMC Microbiol,2010,10:25.
[8]LI J Y,MA Y,HU C Q,et al.Dissemination of cefotaxime-M-producing Escherichia coli isolates in poultry farms,but not swine farms,in China[J].Foodborne Pathog Dis,2010,7(11):1387-1392.
[9]RAO L L,LYU L C,ZENG Z L,et al.Increasing prevalence of extended-spectrum cephalosporin-resistant Escherichia coli in food animals and the diversity of CTX-M genotypes during 2003-2012[J].Vet Microbiol,2014,172(3/4):534-541.
[10]XU G F,AN W,WANG H D,et al.Prevalence and characteristics of extended-spectrum β-lactamase genes in Escherichia coli isolated from piglets with post-weaning diarrhea in Heilongjiang province,China[J].Front Microbiol,2015,6:1103.
[11]中华人民共和国国家卫生和计划生育委员会,国家食品药品监督管理总局.食品安全国家标准 食品微生物学检验 致泻大肠埃希氏菌检验:GB 4789.6—2016[S].北京:中国标准出版社,2016.
[12]Clinical Laboratory Standards Institute. Methods for antimicrobial dilution and disk susceptibility testing of infrequently isolated or fastidious bacteria; approved guideline[S]. Wayne Pennsylvania: Clinical Laboratory Standards Institute, 2015.
[13]XU L, ENSOR V M, GOSSAIN S, et al. Rapid and simple detection of blaCTX-M genes by multiplex PCR assay[J].J Med Microbiol, 2005, 54(pt 12):1183-1187.
[14]ECKERT C, GAUTIER V, SALADIN-ALLARD M, et al. Dissemination of CTX-M-type beta-lactamases among clinical isolates of Enterobacteriaceae in Paris, France[J]. Antimicrob Agents Chemother, 2004, 48(4):1249-1255.
[15]JIANG X F, ZHANG Z, LI M, et al. Detection of extended-spectrum β-lactamases in clinical isolates of Pseudomonas aeruginosa[J]. Antimicrob Agents Chemother 2006, 50(9):2990-2995.
[16]KARCZMARCZYK M, ABBOTT Y, WALSH C, et al. Characterization of multidrug-resistant Escherichia coli isolates from animals presenting at a university veterinary hospital[J]. Appl Environ Microbiol,2011, 77(20): 7104-7112.
[17]BAUERNFEIND A,JUNGWIRTH R,SCHWEIGHART S,et al.Antibacterial activity and beta-lactamase stability of eleven oral cephalosporins[J].Infection,1990,18(3):155-167.
[18]XIA S,FAN X,HUANG Z G,et al.Dominance of CTX-M-type extended-spectrum β-lactamase(ESBL)-producing Escherichia coli isolated from patients with community-onset and hospital-onset infection in China[J].PLoS One,2014,9(7):e100707.
[19]HOANG P H,AWASTHI S P,DO NGUYEN P,et al.Antimicrobial resistance profiles and molecular characterization of Escherichia coli strains isolated from healthy adults in Ho Chi Minh City,Vietnam[J].J Vet Med Sci,2017,79(3):479-485.
[20]黄耀凌,刘智宏,白玉惠,等.恩诺沙星注射液在猪体内的残留消除研究[J].中国兽药杂志,2017,51(3):48-54.
[21]丁丽玲,鲁晓旭,李银生,等.蚯蚓体内恩诺沙星残留的检测方法研究[J].农业环境科学学报,2016,3(2):403-408.
[22]王影,李艳然,韩镌竹,等.多粘菌素耐药性的研究进展[J].微生物学通报,2017,44(1):200-206.
[23]刘艳红,吕淑霞,李颖,等.大肠杆菌耐药性及氟苯尼考耐药基因floR的研究[J].科技学术,2017,53(1):115-118.
[24]XIA L,LIU Y, XIA S,et al.Prevalence of ST1193 clone and IncI1/ST16 plasmid in E-coli isolates carrying blaCTX-M-55 gene from urinary tract infections patients in China[J].Sci Rep,2017,7:44866.
[25]LYU L C,PARTRIDGE S R,HE L Y,et al.Genetic characterization of IncI2 plasmids carrying blaCTX-M-55 spreading in both pets and food animals in China[J].Antimicrob Agents Chemother,2013,57(6):2824-2827.
[26]CARATTOLI A.Plasmids in Gram negatives:molecular typing of resistance plasmids[J].Int J Med Microbiol,2011,301(8):654-658.
[27]MORGAN-LINNELL S K,BOYD L B,STEFFEN D,et al.Mechanisms accounting for fluoroquinolone resistance in Escherichia coli clinical isolates[J].Antimicrob Agents Chemother,2009,53(1):235-241.
[28]HANSEN L H,JOHANNESEN E,BURMLLE M,et al.Plasmid-encoded multidrug efflux pump conferring resistance to olaquindox in Escherichia coli[J].Antimicrob Agents Chemother,2004,48(9):3332-3337.
[29]ENNE V I,LIVERMORE D M,STEPHENS P,et al.Persistence of sulphonamide resistance in Escherichia coli in the UK despite national prescribing restriction[J].Lancet,2001,357(9265):1325-1328.
[30]AJIBOYE R M,SOLBERG O D,LEE B M,et al.Global spread of mobile antimicrobial drug resistance determinants in human and animal Escherichia coli and Salmonella strains causing community-acquired infections[J].Clin Infect Dis,2009,49(3):365-371.
[31]GUERRA B,JUNKER E,SCHROETER A,et al.Phenotypic and genotypic characterization of antimicrobial resistance in German Escherichia coli isolates from cattle,swine and poultry[J].J Antimicrob Chemother,2003,52(3):489-492.
[32]MOMTAZ H,RAHIMI E,MOSHKELANI S.Molecular detection of antimicrobial resistance genes in E.coli isolated from slaughtered commercial chickens in Iran[J].Vet Med(Praha),2012,57(4):193-197.
[33]NSOFOR C A,IROEGBU C U,CALL D R,et al.Detection of antibiotic resistance genes of Escherichia coli from domestic livestock in southeast Nigeria with DNA microarray[J].J Cell Anim Biol,2013,7(12):149-163.
[34]ZHANG F Y,HUO S Y,LI Y R,et al.A survey of the frequency of aminoglycoside antibiotic-resistant genotypes and phenotypes in Escherichia coli in broilers with septicaemia in Hebei,China[J].Br Poul Sci,2014,55(3):305-310.
[35]杜向党,阎若潜,沈建忠.氯霉素类药物耐药机制的研究进展[J].动物医学进展,2004,25(2):27-29.
[36]郑朝朝,刘聚祥,李辉,等.鸡源,猪源大肠杆菌氟苯尼考耐药基因floR的克隆与序列分析[J].华北农学报,2010,25(5):72-75.
[37]YAGUCHI K,OGITANI T,OSAWA R,et al.Virulence factors of avian pathogenic Escherichia coli strains isolated from chickens with colisepticemia in Japan[J].Avian Dis,2007,51(3):656-662.
 王伟,胡豫杰,徐进,彭子欣,李孟寒,闫韶飞,甘辛,董银苹,白瑶,李凤琴.产CTX-M-55型超广谱β-内酰胺酶大肠埃希菌全基因组序列耐药和毒力特征研究[J].中国食品卫生杂志,2017,29(5):519-525.
王伟,胡豫杰,徐进,彭子欣,李孟寒,闫韶飞,甘辛,董银苹,白瑶,李凤琴.产CTX-M-55型超广谱β-内酰胺酶大肠埃希菌全基因组序列耐药和毒力特征研究[J].中国食品卫生杂志,2017,29(5):519-525.